Index >
Results for CNB 134270_18-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 CA-AACTAATCAAAGCGCCCATCCAACACCGACGCCCGCATCAC-TAACATTACATAAAA
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 AA-AACTAATAAAATCGCCCATCCAACACTGATGTCAATATTACTTTAAATTACACAAAA
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 AAGACCTTATCAGGAC-AGCATCCAACTCTGATG-CTTCCTCAGCAGCACAGAATCAAAT
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 AA-AACTAATAAAATCGCCCATCCAACACTGATGTCAATATTACTTTAAATTACACGAAA
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 AA-AACTAATAAAATCGCCCATCCAACACTGATGTCAATATTACTTTAAATTACACGAAA
* * ** ** * * ******** * ** * * * * * **
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 TCAAATTCATTTTTAATTAGGCTAGCAAATTAATACGCAACAGCTGAGGAGTTTAATTAC
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 TCAAATTTATTTTTAATTA-GC----AAATTAATAGGCGGCAGTTGAGGGTTTTAATTAC
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 TC-ATTTTTTAATTAGGCG-GCT---AAATTAATAGGCGGCAGTTTAGGAGTTTAATTAC
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 TCAAATTTATTTTTAATTA-GC----AAATTAATAGGCGGCAGTTGAGGGTTTTAATTAC
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 TCAAATTTATTTTTAATTA-GC----AAATTAATAGGCGGCAGTTGAGGGTTTTAATTAC
** * ** * *** ** ********* ** *** * *** *********
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 TCAATTAACTTCACGCGCGTTAATTTTTAACTATCTAAATTAAGTTGCACATTATTCGAT
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 TCAATTAACTTCACGCAAGTTAATTTTTAACTATCTAAATTAACTTGCGCATTACTCGAT
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 TCAATTAACTTCACGCACGTTAATTTTTAACTATCTAAATTAGGTTGCACATTATTCGAT
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 TCAATTAACTTCACGCAAGTTAATTTTTAACTATCTAAATTAACTTGCACATTACTCGAT
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 TCAATTAACTTCACGCAAGTTAATTTTTAACTATCTAAATTAACTTGCACATTACTCGAT
**************** ************************ **** ***** *****
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 TTGATGATAATTATAGAATTAAATCTAAATTAATTGTTGTGGATGATTTG-------ATA
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 TTGCTGATAATTACAGAATTAAATCTAAATTAATTGTTGGAGGTGATTTGATCCGCTGCT
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 TTGATGATAATTATAGAATTAAATCAAAATTAATTGTTTTGGATGATTTG-------GGT
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 TTGCTGATAATTACAGAATTAAATCTAAATTAATTGTTGGAGGTGATTTGATCCGCTGCT
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 TTGCTGATAATTACAGAATTAAATCTAAATTAATTGTTGGAGGTGATTTGATCCGCTGCT
*** ********* *********** ************ * *******
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 CGACGAGATGCCGCTCCAATTAAAGAAAAAGACGTGGCA----------CTACGGTGTTT
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 GAACTGGATGAGATTCCAATTAACCAGCAAAGAGATTTT----------GT-TGATGTTT
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 GAACTAGATGCAGTTCCAATTAAACATTGAGGGGGGGTTGAAAAAGCCAGT-TGCCATTT
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 GAACTGGATGCAATTCCAATTAACCAGCCAAGAGATTTT----------GT-TGATGTTT
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 GAACTGGATGCAATTCCAATTAACCAGCCAAGAGATTTT----------GT-TGATGTTT
** **** ********* * * * * * ***
134270_18-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 80.55
Mean single sequence MFE: -63.24
Consensus MFE: -34.08
Energy contribution: -35.72
Covariance contribution: 1.64
Mean z-score: -2.14
Structure conservation index: 0.54
SVM decision value: 0.53
SVM RNA-class probability: 0.771469
Prediction: RNA
######################################################################
>134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687
CAAACUAAUCAAAGCGCCCAUCCAACACCGACGCCCGCAUCACUAACAUUACAUAAAAUCAAAUUCAUUUUUAAUUAGGCUAGCAAAUUAAUACGCAACAGCUGAGGAGUUUAAUUACUCAAUUAACUUCACGCGCGUUAAUUUUUAACUAUCUAAAUUAAGUUGCACAUUAUUCGAUUUGAUGAUAAUUAUAGAAUUAAAUCUAAAUUAAUUGUUGUGGAUGAUUUGAUACGACGAGAUGCCGCUCCAAUUAAAGAAAAAGACGUGGCACUACGGUGUUU
.......................(((((((.((..((.((((....((((.(((...((((((((.....((((((.(.....).))))))...(((((.((((.((((((.((((....)))))))))).)).)).(((((((..........)))))))))))).........))))))))((((((((((((......))))...)))))))))))))))...)))).)).))((.((((((((..............)).)))))))).))))))). ( -52.84)
>134270_ENSG00000164853_HUMAN_5374_7839/1-2687
AAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACAAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCGCAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGAGAUUCCAAUUAACCAGCAAAGAGAUUUUGUUGAUGUUU
.(((((((((((((((((..(((((((.((((..........................))))((((((..(((((((....(((((.....(((((((((((((..((((((....))))))..)).(((((((((((((..........))))))))))))).....))))).)))))).)))))....)))))))..))))))...)))))))))))))))(((((..(((((((.((((......)))).))...)))))..).)))).)))))..)))) ( -66.47)
>134270_SINFRUG00000149320_FUGU_3315_5852/1-2687
AAGACCUUAUCAGGACAGCAUCCAACUCUGAUGCUUCCUCAGCAGCACAGAAUCAAAUUCAUUUUUUAAUUAGGCGGCUAAAUUAAUAGGCGGCAGUUUAGGAGUUUAAUUACUCAAUUAACUUCACGCACGUUAAUUUUUAACUAUCUAAAUUAGGUUGCACAUUAUUCGAUUUGAUGAUAAUUAUAGAAUUAAAUCAAAAUUAAUUGUUUUGGAUGAUUUGGGUGAACUAGAUGCAGUUCCAAUUAAACAUUGAGGGGGGGUUGAAAAAGCCAGUUGCCAUUU
............((.((((..............(((((((((((((...((((...)))).........((((..((..((((((((..((((.((((...((((......))))....)))).).)))..))))))))....))..)))).....))))).........((((((((..((....))..)))))))).........(((((((((((((((((.....)))))).))..))))...))))).))))))))((((.....)))).)))))).... ( -66.80)
>134270_ENSRNOG00000001284_RAT_5040_7483/1-2687
AAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACGAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUAACCAGCCAAGAGAUUUUGUUGAUGUUU
.(((((((((((((((((..(((((((.((((.((....................)).))))((((((..(((((((....(((((.....(((((((((((((..((((((....))))))..))..((((((((((((..........))))))))))))......))))).)))))).)))))....)))))))..))))))...))))))))))..........((((...((.((((......)))).))...)))).....))))))))))..)))) ( -65.05)
>134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687
AAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACGAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUAACCAGCCAAGAGAUUUUGUUGAUGUUU
.(((((((((((((((((..(((((((.((((.((....................)).))))((((((..(((((((....(((((.....(((((((((((((..((((((....))))))..))..((((((((((((..........))))))))))))......))))).)))))).)))))....)))))))..))))))...))))))))))..........((((...((.((((......)))).))...)))).....))))))))))..)))) ( -65.05)
>consensus
AA_AACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACAAAAUCAAAUUUAUUUUUAAUUA_GC____AAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUAACCAGCAAAGAGAUUUU__________GU_UGAUGUUU
...........((((((((..((((((...((((....)))).............................................(((((((...(((((((((((((..((((((....))))))..))..((((((((((((..........))))))))))))......))))).)))))).(((((....))))).......)))))))))))))))))))))........((((((.((((......)))).))...))))................................ (-34.08 = -35.72 + 1.64)
134270_18-rev.rnaz
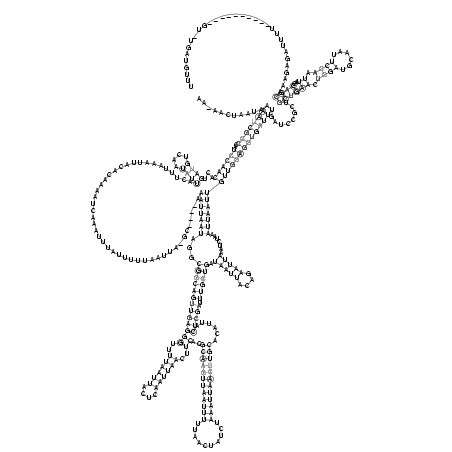
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004