Index >
Results for CNB 234029_27-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
234029_ENSMUSG00000045374_MOUSE_1538_4188/1-2892 AAATCA-TCTTCTCCTG---TCCCAGGCCTATCTCTAGTCCAGCCAGTCTCACATGGAGT
234029_ENSRNOG00000003243_RAT_1679_4322/1-2892 AAGTCA-TCTTCCCCTG---CCGTGGGCCTCTCTCTAGTCCAGCCACTCTCACATGGAGT
234029_ENSDARG00000018072_ZEBRAFISH_4044_6930/1-2892 GAGCCACTTAAGTCCAGAGCTTCTATTGCTTGCTCAAGCTCTTCATCCCGGCCTACTACT
234029_ENSG00000167716_HUMAN_7345_10000/1-2892 AAGTCG-TCTTCTCCTG---CCACCGGCCTGTCTCTGGTCCAGCTAGCCTCACAGGGAGT
* * * ** * ** *** * * * * * * *
234029_ENSMUSG00000045374_MOUSE_1538_4188/1-2892 GGCC-TCTACGACAAGC-CTGCCCGCACCATTTATAAGTTGTC------------CTGAA
234029_ENSRNOG00000003243_RAT_1679_4322/1-2892 GACC-TCTACAACATGC-CTGCCCGCACCATTAATAAGTTGTC------------CTGAG
234029_ENSDARG00000018072_ZEBRAFISH_4044_6930/1-2892 GACCAGCCACTAGATTCACACCCTGTTGCCTCTGGGACTAATCCATCCACGCCGTCCATT
234029_ENSG00000167716_HUMAN_7345_10000/1-2892 GGCC-TCTAAAACTGGC-CGGCCCACTCCATTTGGAAGCTGTC---CCGGGTTTTCCGTG
* ** * * * * * ** * * * ** *
234029_ENSMUSG00000045374_MOUSE_1538_4188/1-2892 ATGTCCTCCTGGCCTGTGGCCTCCCGAATAGGCTGGACCAACAGCCGGGGGATGAGTGGA
234029_ENSRNOG00000003243_RAT_1679_4322/1-2892 ATGTCCTCCTGGCCCGTGGCCTCCTGAATAGGCTGGACCAACAGCCGGGGTATGAGTGGA
234029_ENSDARG00000018072_ZEBRAFISH_4044_6930/1-2892 GTGGTTTCAAGGGGTGGAATCTGCC--ATGTTGT-AACATTGACACGTCCAAAGTGAGGA
234029_ENSG00000167716_HUMAN_7345_10000/1-2892 AAGTCCTCCCGGCCTGTGGTCTCCTGGATGGTCTGGACCAACAGCTTGGGGATGAGGGGA
* ** ** * ** * ** * ** * * * * ***
234029_ENSMUSG00000045374_MOUSE_1538_4188/1-2892 GGTTCAGGGGCCAGGGCAGGAGATCCAGCCAGGCGTTGGGGGTGTGGCTGATCAAATAGC
234029_ENSRNOG00000003243_RAT_1679_4322/1-2892 GGTTCAGGGGCCAGGGCAGGAGCCCCAGCCAGGCGTTGGGGGTGTGGCTGATCAAACAGC
234029_ENSDARG00000018072_ZEBRAFISH_4044_6930/1-2892 GGTTCTGGAGGTGCATACTGGTAAAGGGCAAGACGAGGTGGGTGGGGGTGGTCAAACAGT
234029_ENSG00000167716_HUMAN_7345_10000/1-2892 GGCTCGGGGGCAAGGGCAGGAGCCCCAGCCAGGCGCTGGGGGTGTGGCTGATCGAAGAGC
** ** ** * * ** ** ** * ***** ** ** ** ** **
234029_ENSMUSG00000045374_MOUSE_1538_4188/1-2892 TGTACCACGCCATAGCTGGTCAGATGGGTGTGAGCGTCCACCAGGTGCAGACACACATTC
234029_ENSRNOG00000003243_RAT_1679_4322/1-2892 TGTACCACGCCATAGCTGGTCAGATGGGTGTGAGCATCCACCAGGTGCAGACACACATTC
234029_ENSDARG00000018072_ZEBRAFISH_4044_6930/1-2892 TGCACCACTCCGTAGCTGGTGAGATGAGTGTGGTTATCTACCAAGTGCAGGCAGACATTC
234029_ENSG00000167716_HUMAN_7345_10000/1-2892 TGCACCACCCCGTAGCTGGCCAGGTGAGTGTGGGCGTCCACCAGGTGCAGACACACATTC
** ***** ** ******* ** ** ***** ** **** ****** ** ******
234029_27-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 66.95
Mean single sequence MFE: -118.98
Consensus MFE: -62.83
Energy contribution: -63.51
Covariance contribution: 0.69
Mean z-score: -1.55
Structure conservation index: 0.53
SVM decision value: 0.47
SVM RNA-class probability: 0.750696
Prediction: RNA
######################################################################
>234029_ENSMUSG00000045374_MOUSE_1538_4188/1-2892
AAAUCAUCUUCUCCUGUCCCAGGCCUAUCUCUAGUCCAGCCAGUCUCACAUGGAGUGGCCUCUACGACAAGCCUGCCCGCACCAUUUAUAAGUUGUCCUGAAAUGUCCUCCUGGCCUGUGGCCUCCCGAAUAGGCUGGACCAACAGCCGGGGGAUGAGUGGAGGUUCAGGGGCCAGGGCAGGAGAUCCAGCCAGGCGUUGGGGGUGUGGCUGAUCAAAUAGCUGUACCACGCCAUAGCUGGUCAGAUGGGUGUGAGCGUCCACCAGGUGCAGACACACAUUC
...((((((((((((((((..((((........(((..((((.(((.....))).))))......)))...((((.((...((((((((..(((.......)))...((((((((.(((((((..((.....))..)).)).))))))))))))))))))).))..)))))))).)))))))))).(((((..(((((....((((..((((......))))..)))))))))...)))))..))))))(((((.(((.((....)))))...))))).... ( -118.00)
>234029_ENSRNOG00000003243_RAT_1679_4322/1-2892
AAGUCAUCUUCCCCUGCCGUGGGCCUCUCUCUAGUCCAGCCACUCUCACAUGGAGUGACCUCUACAACAUGCCUGCCCGCACCAUUAAUAAGUUGUCCUGAGAUGUCCUCCUGGCCCGUGGCCUCCUGAAUAGGCUGGACCAACAGCCGGGGUAUGAGUGGAGGUUCAGGGGCCAGGGCAGGAGCCCCAGCCAGGCGUUGGGGGUGUGGCUGAUCAAACAGCUGUACCACGCCAUAGCUGGUCAGAUGGGUGUGAGCAUCCACCAGGUGCAGACACACAUUC
...((((((...(((((((.((((.((((...((..((((((((((.....))))))............(((......)))..........))))..)))))).)))).)(((((((.((((((((...((((((((......)))))....)))....))))).))).)))))))))))))....(((((..(((((....((((..((((......))))..)))))))))...)))))..))))))(((((.(((((.....)))))...))))).... ( -120.30)
>234029_ENSDARG00000018072_ZEBRAFISH_4044_6930/1-2892
GAGCCACUUAAGUCCAGAGCUUCUAUUGCUUGCUCAAGCUCUUCAUCCCGGCCUACUACUGACCAGCCACUAGAUUCACACCCUGUUGCCUCUGGGACUAAUCCAUCCACGCCGUCCAUUGUGGUUUCAAGGGGUGGAAUCUGCCAUGUUGUAACAUUGACACGUCCAAAGUGAGGAGGUUCUGGAGGUGCAUACUGGUAAAGGGCAAGACGAGGUGGGUGGGGGUGGUCAAACAGUUGCACCACUCCGUAGCUGGUGAGAUGAGUGUGGUUAUCUACCAAGUGCAGGCAGACAUUC
...........(((.((.....))..(((((((.(..((((.((.(((((((.(((.......((.(((((((((((.((((((...(((.(..((((...............))))...).))).....))))))))))))(((...((((.....((.(((.((((.((........)).)))).))))).....))))..))).......))))).))(((((((((((....))).)))))))))))))))).)))).)))).((((.....)))).).)))))))))).... ( -101.46)
>234029_ENSG00000167716_HUMAN_7345_10000/1-2892
AAGUCGUCUUCUCCUGCCACCGGCCUGUCUCUGGUCCAGCUAGCCUCACAGGGAGUGGCCUCUAAAACUGGCCGGCCCACUCCAUUUGGAAGCUGUCCCGGGUUUUCCGUGAAGUCCUCCCGGCCUGUGGUCUCCUGGAUGGUCUGGACCAACAGCUUGGGGAUGAGGGGAGGCUCGGGGGCAAGGGCAGGAGCCCCAGCCAGGCGCUGGGGGUGUGGCUGAUCGAAGAGCUGCACCACCCCGUAGCUGGCCAGGUGAGUGUGGGCGUCCACCAGGUGCAGACACACAUUC
..((.((((.(.((((.((((((((..((((((...............))))))..))))........((((((((....(((....))).(((..((((((((((((.(...((((.((.(((.((((((((((.....))...))))).)))))).)))))).).))))))))))))(((..((((....))))..))).)))((.(((((((..(((........)))..)))).))).)).))))))))))))...((((....)))))))).).))))))...... ( -136.16)
>consensus
AAGUCA_UCUUCUCCUG___CCCCAGGCCUAUCUCUAGUCCAGCCACCCUCACAUGGAGUGACC_UCUACAACAUGC_CUGCCCGCACCAUUUAGAAGUUGUC____________CCGAGAUGUCCUCCUGGCCUGUGGCCUCCCGAAUAGGCUGGACCAACAGCCGGGGGAUGAGUGGAGGUUCAGGGGCCAGGGCAGGAGACCCAGCCAGGCGUUGGGGGUGUGGCUGAUCAAACAGCUGCACCACGCCAUAGCUGGUCAGAUGAGUGUGAGCAUCCACCAGGUGCAGACACACAUUC
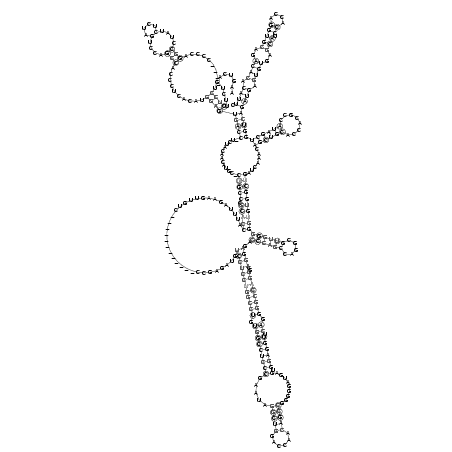
...........((((..........(((....(....)....)))..........))))(((((..............(((((.(((((..................................((((((((((((.(((((((((.....(((((......))))).........).)))))).)).))))))))..))))..((((((.....)))))))))))))))).......((((((........)))))))))))((((.((((..(((.((....)))))..)))).)))). (-62.83 = -63.51 + 0.69)
234029_27-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004