Index >
Results for CNB 268475-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
268475_ENSG00000171532_HUMAN_8474_8575/1-108 GGGG-----TACCAGCCTCTATGCCAGG-CCATATGGGCTTGGCACGTCACGGGCAGCAG
268475_ENSRNOG00000028417_RAT_8609_8710/1-108 GGGG-----TACCAGCCTCTATGCCAGG-CCATATGGGCTTGGCACGTCACGGGCAGCAG
268475_ENSDARG00000016854_ZEBRAFISH_8451_8554/1-108 AGGGTTACATACCTGCTCTTATGCCAAG-CCATATGG--TGGGCACGTCACTGGCAGGAG
268475_ENSMUSG00000038255_MOUSE_8602_8703/1-108 GGGG-----TACCAGCCTCTATGCCAGG-CCATATGGGCTTGGCACGTCACGGGCAGCAG
268475_SINFRUG00000148338_FUGU_7962_8066/1-108 -GGGTTACATACCAGCTCTGATGCCAGGCCCATATGG--CTGGCACGTCATTGGCAAGAG
*** **** ** ****** * ******** ********* **** **
268475_ENSG00000171532_HUMAN_8474_8575/1-108 CTATCACATGAGAGACGGTGATTGGCATGCGTCTGCACTGGGGGAGAG
268475_ENSRNOG00000028417_RAT_8609_8710/1-108 CTATCACATGAGAGACGGTGATTGGCATGCGTCTGCATTGGGGGAGAG
268475_ENSDARG00000016854_ZEBRAFISH_8451_8554/1-108 CCATCACATGAGAGCGGGTGATTGACACGCGCTCGCATTCGTAGAGA-
268475_ENSMUSG00000038255_MOUSE_8602_8703/1-108 CTATCACATGAGAGACGGTGATTGGCATGCGTCTGCATTGGGGGAGAG
268475_SINFRUG00000148338_FUGU_7962_8066/1-108 CCATCACATGAGAGCCGGTGATTGACATGCGGCCGCATTCACAGAGAC
* ************ ******** ** *** *** * ****
268475-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 108
Mean pairwise identity: 82.21
Mean single sequence MFE: -40.26
Consensus MFE: -28.44
Energy contribution: -27.72
Covariance contribution: -0.72
Mean z-score: -1.64
Structure conservation index: 0.71
SVM decision value: 0.14
SVM RNA-class probability: 0.602184
Prediction: RNA
######################################################################
>268475_ENSG00000171532_HUMAN_8474_8575/1-108
GGGGUACCAGCCUCUAUGCCAGGCCAUAUGGGCUUGGCACGUCACGGGCAGCAGCUAUCACAUGAGAGACGGUGAUUGGCAUGCGUCUGCACUGGGGGAGAG
((....))..(((((((((((((((.....))))))))).....(((((.(((((((((((.(....)...)))).)))).))))))))...)))))).... ( -41.80)
>268475_ENSRNOG00000028417_RAT_8609_8710/1-108
GGGGUACCAGCCUCUAUGCCAGGCCAUAUGGGCUUGGCACGUCACGGGCAGCAGCUAUCACAUGAGAGACGGUGAUUGGCAUGCGUCUGCAUUGGGGGAGAG
((....))..(((((((((((((((.....))))))))).....(((((.(((((((((((.(....)...)))).)))).))))))))...)))))).... ( -41.90)
>268475_ENSDARG00000016854_ZEBRAFISH_8451_8554/1-108
AGGGUUACAUACCUGCUCUUAUGCCAAGCCAUAUGGUGGGCACGUCACUGGCAGGAGCCAUCACAUGAGAGCGGGUGAUUGACACGCGCUCGCAUUCGUAGAGA
...((((..((((((((((((((((..(((....))).)))..((...((((....))))..)))))))))))))))..))))..((....))........... ( -37.30)
>268475_ENSMUSG00000038255_MOUSE_8602_8703/1-108
GGGGUACCAGCCUCUAUGCCAGGCCAUAUGGGCUUGGCACGUCACGGGCAGCAGCUAUCACAUGAGAGACGGUGAUUGGCAUGCGUCUGCAUUGGGGGAGAG
((....))..(((((((((((((((.....))))))))).....(((((.(((((((((((.(....)...)))).)))).))))))))...)))))).... ( -41.90)
>268475_SINFRUG00000148338_FUGU_7962_8066/1-108
GGGUUACAUACCAGCUCUGAUGCCAGGCCCAUAUGGCUGGCACGUCAUUGGCAAGAGCCAUCACAUGAGAGCCGGUGAUUGACAUGCGGCCGCAUUCACAGAGAC
.(((.....)))..(((((((((..(((((((((.((((((.(.((((((((....))))....))))).)))))).))....))).))))))))...))))).. ( -38.40)
>consensus
GGGG_____UACCAGCCUCUAUGCCAGG_CCAUAUGGGCUUGGCACGUCACGGGCAGCAGCUAUCACAUGAGAGACGGUGAUUGGCAUGCGUCUGCAUUGGGGGAGAG
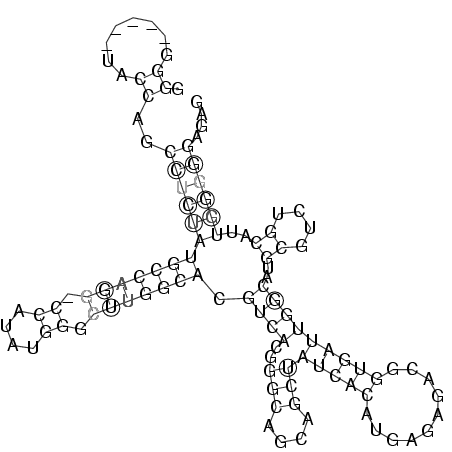
.((........))..(((((((((((((.((....)).))))))).((((..(((....)))(((((..........)))))))))..((....))..)))))).... (-28.44 = -27.72 + -0.72)
268475-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004