Index >
Results for CNB 300319_1-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
300319_SINFRUG00000145103_FUGU_7631_7985/1-365 TTTAAATCTGATTATCAAATTAGGAGACTTTGAGACAAATCCTCTTAGATCTGATACTGT
300319_ENSG00000108001_HUMAN_7069_7424/1-365 TTTAAATCTGATTACCAAATTAGAAGGTTTAAAAACAAATCCTTCTAAATCTGATACTGT
300319_ENSMUSG00000010476_MOUSE_7037_7393/1-365 TTTAAATCTGATTACCAAATTAGAAGGTTTAAAAGCAAATCCTTCTAAATCTGATACTGT
300319_ENSDARG00000003613_ZEBRAFISH_7458_7781/1-365 TTTCAATCTGATTACCAAATTAG--GGTTTTAGTCTAAATCCCTTTAAATCTGATACTGT
*** ********** ******** * ** ****** ** ************
300319_SINFRUG00000145103_FUGU_7631_7985/1-365 TGACTGGAGA--GG-GA-AAATGGATCTCCACAGCCGTCACGT-AGGTAACGATGCTG-T
300319_ENSG00000108001_HUMAN_7069_7424/1-365 TGACTAAAGA--GGNNN-NNNNNGTTCTATAAGCCCATCACATAAGGTAACGATCCTGCC
300319_ENSMUSG00000010476_MOUSE_7037_7393/1-365 TGACTAAAGAGGGG-GAGAAAAAGTTCCATAAGCCCATCACATAAGGTAACGATCCTGCC
300319_ENSDARG00000003613_ZEBRAFISH_7458_7781/1-365 TGACT--AGA-----GA-A----GTTCAACAATCCCATCGCGT-AGGTAACGATCCCG-T
***** *** * ** * ** ** * * ********** * *
300319_SINFRUG00000145103_FUGU_7631_7985/1-365 CCTGAAAG----GGAGTTTTAAACCGTATTTGACAACAAATGCAGCTGCCCCGCTATTTC
300319_ENSG00000108001_HUMAN_7069_7424/1-365 CCAGAAAGAAAAGGGGTTTTAAACC-TTTTTGACAACAAATGCAGCTGCCCCACTATTTT
300319_ENSMUSG00000010476_MOUSE_7037_7393/1-365 CCAGAAAGAAAGGGGGTTTTAAACC-TTTTTGACAACAAATGCAGCTGCCCCGCTATTTT
300319_ENSDARG00000003613_ZEBRAFISH_7458_7781/1-365 CCTAAAAA----GGAGTTTTAAACCTTATTTGACAACAAATGCAGCTGCCCCTCTATTTT
** *** ** ********** * ************************ ******
300319_SINFRUG00000145103_FUGU_7631_7985/1-365 TGAAGATATTAGAGGAAAATGAATGCAAATCTGTGTGCAGGAGCCATCTGGACGTGGAGA
300319_ENSG00000108001_HUMAN_7069_7424/1-365 GGACGATATTAAGCGAAAATGAATGCAAATCTGTGTTCAGCAGCCATCTGG-CCCGAAAT
300319_ENSMUSG00000010476_MOUSE_7037_7393/1-365 GGACGATATTAAGCGAAAATGAATGCAAATCTGTGTTCGGCAGCCATCTGG-CCCGAAAT
300319_ENSDARG00000003613_ZEBRAFISH_7458_7781/1-365 GGAAGATATTAGGCGAAAATGAATGCAAATCTGTGTTCAGAAGCCATCTGG-CCGGAGAC
** ******* ********************** * * ********** * *
300319_SINFRUG00000145103_FUGU_7631_7985/1-365 GAGGGAGTCAGCTGCTGCACACACTGAGGCTCGGAGCCTGAATCCGTCTCAGCTCATTCT
300319_ENSG00000108001_HUMAN_7069_7424/1-365 GGGGGAATCAGCTGCT-CTCACA-ACAGGCTCGGAGGCTGAATCCATTTCAGCTCATTTT
300319_ENSMUSG00000010476_MOUSE_7037_7393/1-365 GGGGGAATCAGCTG-T-CTCACA-ACAGGCTCGGGGGCTGAATCCATTTCAGCTCATTTT
300319_ENSDARG00000003613_ZEBRAFISH_7458_7781/1-365 TGGAGAATCAGCTGCT-C-----------------GCCTGAATCCATCTCGGCTCATTTC
* ** ******* * * * ******** * ** *******
300319_1-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 79.29
Mean single sequence MFE: -81.88
Consensus MFE: -43.67
Energy contribution: -47.05
Covariance contribution: 3.38
Mean z-score: -2.28
Structure conservation index: 0.53
SVM decision value: 0.44
SVM RNA-class probability: 0.738632
Prediction: RNA
######################################################################
>300319_SINFRUG00000145103_FUGU_7631_7985/1-365
UUUAAAUCUGAUUAUCAAAUUAGGAGACUUUGAGACAAAUCCUCUUAGAUCUGAUACUGUUGACUGGAGAGGGAAAAUGGAUCUCCACAGCCGUCACGUAGGUAACGAUGCUGUCCUGAAAGGGAGUUUUAAACCGUAUUUGACAACAAAUGCAGCUGCCCCGCUAUUUCUGAAGAUAUUAGAGGAAAAUGAAUGCAAAUCUGUGUGCAGGAGCCAUCUGGACGUGGAGAGAGGGAGUCAGCUGCUGCACACACUGAGGCUCGGAGCCUGAAUCCGUCUCAGCUCAUUCU
.......((((((....))))))(((....((((((...(((((((((.((..........)))).))))))).....((((((((..((((((((.((((((......((..((((....)))))).....))).))).)))).......(((((((((((.((.(((((((.....)))))))..............((..(((.((((.....)))).)))..)).)).))).).)))))))............)))).)))).....)))))))))).)))..... ( -86.40)
>300319_ENSG00000108001_HUMAN_7069_7424/1-365
UUUAAAUCUGAUUACCAAAUUAGAAGGUUUAAAAACAAAUCCUUCUAAAUCUGAUACUGUUGACUAAAGAGGNNNNNNNNGUUCUAUAAGCCCAUCACAUAAGGUAACGAUCCUGCCCCAGAAAGAAAAGGGGUUUUAAACCUUUUUGACAACAAAUGCAGCUGCCCCACUAUUUUGGACGAUAUUAAGCGAAAAUGAAUGCAAAUCUGUGUUCAGCAGCCAUCUGGCCCGAAAUGGGGGAAUCAGCUGCUCUCACAACAGGCUCGGAGGCUGAAUCCAUUUCAGCUCAUUUU
...................(((((((((((......))).)))))))).(((((..((((((..((((((((.............................(((.......)))(((((..........)))))......)))))))).........(((((((.(((.(((((((((..........((.....((((((((....)))))))))).(((....)))))))))))))))...))))))).....))))))..)))))(((((((.....)))))))...... ( -86.00)
>300319_ENSMUSG00000010476_MOUSE_7037_7393/1-365
UUUAAAUCUGAUUACCAAAUUAGAAGGUUUAAAAGCAAAUCCUUCUAAAUCUGAUACUGUUGACUAAAGAGGGGGAGAAAAAGUUCCAUAAGCCCAUCACAUAAGGUAACGAUCCUGCCCCAGAAAGAAAGGGGGUUUUAAACCUUUUUGACAACAAAUGCAGCUGCCCCGCUAUUUUGGACGAUAUUAAGCGAAAAUGAAUGCAAAUCUGUGUUCGGCAGCCAUCUGGCCCGAAAUGGGGGAAUCAGCUGUCUCACAACAGGCUCGGGGGCUGAAUCCAUUUCAGCUCAUUUU
...................(((((((((((......))).))))))))..((((..((((((..((((((((.((((......))))..((((((.........((((.......))))((.........))))))))....)))))))).........(((((((.(((.(((((((((..........((.....((((((((....)))))))))).(((....)))))))))))))))...)))))))....))))))..))))((((((((.....))))))))..... ( -90.10)
>300319_ENSDARG00000003613_ZEBRAFISH_7458_7781/1-365
UUUCAAUCUGAUUACCAAAUUAGGGUUUUAGUCUAAAUCCCUUUAAAUCUGAUACUGUUGACUAGAGAAGUUCAACAAUCCCAUCGCGUAGGUAACGAUCCCGUCCUAAAAAGGAGUUUUAAACCUUAUUUGACAACAAAUGCAGCUGCCCCUCUAUUUUGGAAGAUAUUAGGCGAAAAUGAAUGCAAAUCUGUGUUCAGAAGCCAUCUGGCCGGAGACUGGAGAAUCAGCUGCUCGCCUGAAUCCAUCUCGGCUCAUUUC
...............(((((.(((((((......(((..(((((......(((..((((((((.....)).)))))))))........((((..(((....))))))).)))))..))).)))))))))))).........(((((((...(((((((((((.((((....(((.....((((((((....))))))))...)))))))..))))))..)))))...)))))))..(((.((.......)))))....... ( -65.00)
>consensus
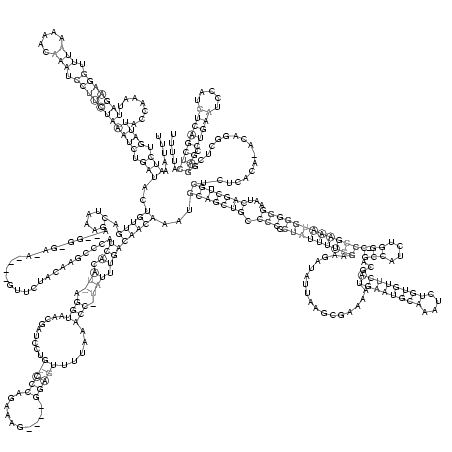
UUUAAAUCUGAUUACCAAAUUAGAAGGUUUAAAAACAAAUCCUUCUAAAUCUGAUACUGUUGACUAAAGA__GG_GA_A____GUUCUACAAGCCCAUCACAU_AGGUAACGAUCCUG_CCCAGAAAG____GGAGUUUUAAACC_UAUUUGACAACAAAUGCAGCUGCCCCGCUAUUUUGGAAGAUAUUAAGCGAAAAUGAAUGCAAAUCUGUGUUCAGCAGCCAUCUGG_CCCGAAAUGGGGGAAUCAGCUGCU_CUCACA_ACAGGCUCGGAGCCUGAAUCCAUCUCAGCUCAUUUU
.....(((.((((.......(((((((.(((.....))).))))))))))).)))..(((((.(....)............................(((.(((.(((.........(((((..........))))).....))).))).))))))))...(((((((.(((.(((((((((.................((((((((....))))))))...(((....)).)))))))))))))...)))))))..................(((.(((((.....))))))))..... (-43.67 = -47.05 + 3.38)
300319_1-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004