Index >
Results for CNB 300387
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
300387_ENSG00000130477_HUMAN_8509_8743/1-236 -CAGGGGCTGCGGAGAAGAGCTCCAAGCACGGGGCGGAGGACCGGACACAGAACATCATC
300387_SINFRUG00000142610_FUGU_8110_8343/1-236 -CAGGAGCTGCTGAGAAGAGTTCCAAGACGGGGGCTGAGGACCGGACACAGCACATCATC
300387_ENSMUSG00000028456_MOUSE_9113_9347/1-236 -TAGGGGCTGCTGAAAAGAGCTCTAAGCATGGAGCTGAGGACCGAACTCAGAACATTATC
300387_ENSRNOG00000008237_RAT_9090_9325/1-236 TCAGGGGCCGCTGAAAAGAGCTCTAAGCACGGAGCTGAGGACCGGACTCAGAACATTATC
300387_ENSDARG00000028370_ZEBRAFISH_9575_9808/1-236 -CAGGGGCGGCAGAAAAGAGCTCTAAACATGGAGCTGAAGATCGCACACAGAACATTATT
*** ** ** ** ***** ** ** ** ** ** ** ** ** *** **** **
300387_ENSG00000130477_HUMAN_8509_8743/1-236 ATGGTGCTCAAGGACCGCATGAAGATCCGGGAGCGCAACAAGCCCGAGATCTTCGAGCTC
300387_SINFRUG00000142610_FUGU_8110_8343/1-236 ATGGCCATGAAGGACAGGATGAAGATCCGCGAGAGGAACAAGCCGGAGATCTTCGAGGAG
300387_ENSMUSG00000028456_MOUSE_9113_9347/1-236 ATGGCCATGAAGGACCGTATGAAGATCCGAGAACGGAATAAACCAGAGATCTTTGAGGTT
300387_ENSRNOG00000008237_RAT_9090_9325/1-236 ATGGCCATGAAGGACCGTATGAAGATCCGAGAACGGAATAAACCAGAGATCTTTGAGGTT
300387_ENSDARG00000028370_ZEBRAFISH_9575_9808/1-236 ATGGTGCTAAAAGACCGCATGAAGATCCGTGAACGGAATAAGCCTGAGATATTTGAGTTG
**** * ** *** * *********** ** * ** ** ** ***** ** ***
300387_ENSG00000130477_HUMAN_8509_8743/1-236 ATCCAGGAGATCTTCGCGGTGACCAAGACGGCGCACACGCAGCAGATGAAGGCGGTCAAG
300387_SINFRUG00000142610_FUGU_8110_8343/1-236 ATCCGGAGGATCTTCAACATCACCAAGATGATCCACGTCCAACACATGAAGAGCATCAAA
300387_ENSMUSG00000028456_MOUSE_9113_9347/1-236 ATCCGGGATGTGTTCACAGTGAGCAAAGTTGCGCATGTGCAGCAGATGAAAACAGTAAAG
300387_ENSRNOG00000008237_RAT_9090_9325/1-236 ATCCGGGATGTGTTCACAGTGAGCAAAGTTGCCCACGTGCAGCAGATGAAAACAGTAAAG
300387_ENSDARG00000028370_ZEBRAFISH_9575_9808/1-236 ATTCAGGATGTGTTTGCAGTCATTAAGGCCACACATGTGGCACAGATGAAGCAGATTAAG
** * * * ** * * ** ** ** ***** * **
300387_ENSG00000130477_HUMAN_8509_8743/1-236 CAGAGCGTGCTGGACGGCACGTCCAAGTGGTCCGCCAAGATCAGCATCACCGGTGA
300387_SINFRUG00000142610_FUGU_8110_8343/1-236 CAAAATGTCCTCGACGGCTCGTCCAAGTGGTCGGCTAAGATTGCCATCAATGGTG-
300387_ENSMUSG00000028456_MOUSE_9113_9347/1-236 CAGAGTGTCCTGGATGGCACCTCCAAGTGGTCAGCCAAAATTACCATCACGGGTAA
300387_ENSRNOG00000008237_RAT_9090_9325/1-236 CAGAGTGTCCTGGATGGCACCTCCAAGTGGTCGGCCAAAATTACCATCACGGGTAA
300387_ENSDARG00000028370_ZEBRAFISH_9575_9808/1-236 CAAAGTGTCCTGGATGGAACGTCTAAGTGGTCGGCCAAGATCAGCATCACCGGTA-
** * ** ** ** ** * ** ******** ** ** ** ***** ***
//
300387.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 236
Mean pairwise identity: 77.52
Mean single sequence MFE: -79.48
Consensus MFE: -36.62
Energy contribution: -36.60
Covariance contribution: -0.02
Mean z-score: -1.84
Structure conservation index: 0.46
SVM decision value: -0.01
SVM RNA-class probability: 0.528351
Prediction: RNA
######################################################################
>300387_ENSG00000130477_HUMAN_8509_8743/1-236
CAGGGGCUGCGGAGAAGAGCUCCAAGCACGGGGCGGAGGACCGGACACAGAACAUCAUCAUGGUGCUCAAGGACCGCAUGAAGAUCCGGGAGCGCAACAAGCCCGAGAUCUUCGAGCUCAUCCAGGAGAUCUUCGCGGUGACCAAGACGGCGCACACGCAGCAGAUGAAGGCGGUCAAGCAGAGCGUGCUGGACGGCACGUCCAAGUGGUCCGCCAAGAUCAGCAUCACCGGUGA
..((.((((((..(((((.((((.(((((.(..(((....)))..)......(((....))))))))...(((..((.((((((((((((...........)))).)))))))).))...))).)))).)))))(((.((.......)).)))...)))))).((((..((((((((....(..((((((....))))))..)...))).)))))........)))).))..... ( -92.00)
>300387_SINFRUG00000142610_FUGU_8110_8343/1-236
CAGGAGCUGCUGAGAAGAGUUCCAAGACGGGGGCUGAGGACCGGACACAGCACAUCAUCAUGGCCAUGAAGGACAGGAUGAAGAUCCGCGAGAGGAACAAGCCGGAGAUCUUCGAGGAGAUCCGGAGGAUCUUCAACAUCACCAAGAUGAUCCACGUCCAACACAUGAAGAGCAUCAAACAAAAUGUCCUCGACGGCUCGUCCAAGUGGUCGGCUAAGAUUGCCAUCAAUGGUG
...(((((((.......((((((......))))))((((((.(....).((...((.(((((....((..((((.((((((((((((.(....).......(((((..(((....)))..))))).))))))))..((((.....))))))))..))))..))))))).))))............))))))).))))))..(((..((((.(((.......))))))).))).. ( -79.60)
>300387_ENSMUSG00000028456_MOUSE_9113_9347/1-236
UAGGGGCUGCUGAAAAGAGCUCUAAGCAUGGAGCUGAGGACCGAACUCAGAACAUUAUCAUGGCCAUGAAGGACCGUAUGAAGAUCCGAGAACGGAAUAAACCAGAGAUCUUUGAGGUUAUCCGGGAUGUGUUCACAGUGAGCAAAGUUGCGCAUGUGCAGCAGAUGAAAACAGUAAAGCAGAGUGUCCUGGAUGGCACCUCCAAGUGGUCAGCCAAAAUUACCAUCACGGGUAA
....(((((((......(((((((....)))))))((((......((((((...((((.((((((.....)).))))))))...((((....))))..............))))))((((((((((((.(((((.....)))))..((((((....)))))).......................)))))))))))).)))).....)).)))))....(((((......))))) ( -75.30)
>300387_ENSRNOG00000008237_RAT_9090_9325/1-236
UCAGGGGCCGCUGAAAAGAGCUCUAAGCACGGAGCUGAGGACCGGACUCAGAACAUUAUCAUGGCCAUGAAGGACCGUAUGAAGAUCCGAGAACGGAAUAAACCAGAGAUCUUUGAGGUUAUCCGGGAUGUGUUCACAGUGAGCAAAGUUGCCCACGUGCAGCAGAUGAAAACAGUAAAGCAGAGUGUCCUGGAUGGCACCUCCAAGUGGUCGGCCAAAAUUACCAUCACGGGUAA
.....((((.(((.....((((((......)))))).....(((..(((.((.(.((((.((((((.....)).)))))))).).)).)))..))).......))).((((((((((((((((((((((.(((((.....)))))..(((((......))))).......................))))))))))..)))).)))).))))))))....(((((......))))) ( -76.30)
>300387_ENSDARG00000028370_ZEBRAFISH_9575_9808/1-236
CAGGGGCGGCAGAAAAGAGCUCUAAACAUGGAGCUGAAGAUCGCACACAGAACAUUAUUAUGGUGCUAAAAGACCGCAUGAAGAUCCGUGAACGGAAUAAGCCUGAGAUAUUUGAGUUGAUUCAGGAUGUGUUUGCAGUCAUUAAGGCCACACAUGUGGCACAGAUGAAGCAGAUUAAGCAAAGUGUCCUGGAUGGAACGUCUAAGUGGUCGGCCAAGAUCAGCAUCACCGGUA
..((....((.......(((((((....)))))))...((((((((.((...........)))))).....((((((.......((((....)))).........((((.......((.((((((((..(((((((..(((((...(((((....)))))...))))).))))))........)..)))))))).))..))))..))))))......)))).))....)).... ( -74.20)
>consensus
_CAGGGGCUGCUGAAAAGAGCUCUAAGCACGGAGCUGAGGACCGGACACAGAACAUUAUCAUGGCCAUGAAGGACCGCAUGAAGAUCCGAGAACGGAAUAAGCCAGAGAUCUUUGAGGUGAUCCGGGAUGUGUUCACAGUGACCAAGGUGGCCCACGUGCAGCAGAUGAAGACAGUAAAGCAGAGUGUCCUGGAUGGCACGUCCAAGUGGUCGGCCAAGAUUACCAUCACCGGUAA
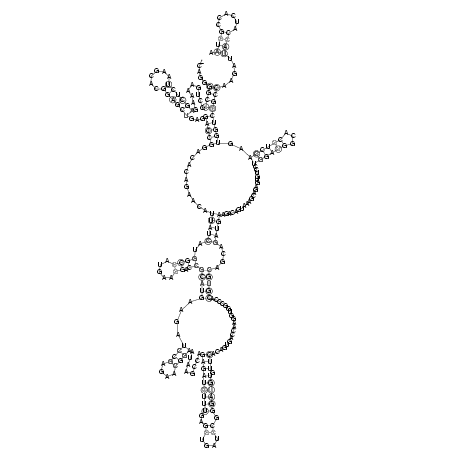
.....((((.........((((((......))))))...(((((...........(((((..((((.....)).))(((((....((((....))))........(((((((((..((....)).)))))).)))....................)))))....))))).....................((((((...))))))..))))))))).....((((......)))). (-36.62 = -36.60 + -0.02)
300387.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004