Index >
Results for CNB 456150-rev
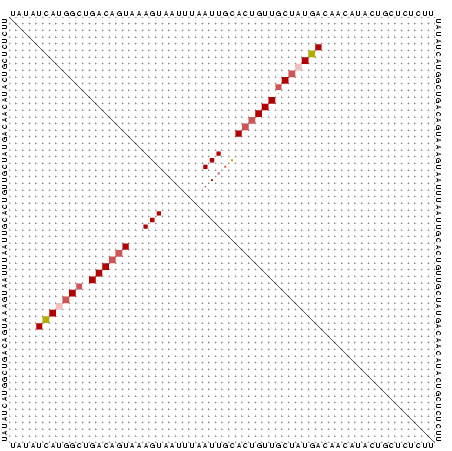
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
456150_ENSG00000053254_HUMAN_2206_2269/1-64 TATATCATGGCTGACAGTAAAGTAATTTAATTGCACTGTTGCTATGACAACATGCTGCTC
456150_ENSMUSG00000033713_MOUSE_7446_7509/1-64 TATATCATGGCTGACAGTAAAGTAATTTAATTGCACTGTTGCTATGACAACATACTGCTC
456150_SINFRUG00000137365_FUGU_7988_8049/1-64 -ACATTACGGCTGACAGTAAAGTCATCTCATTTTACTGTTGCTATGACAACAAACTCCTC
456150_ENSRNOG00000004709_RAT_7742_7803/1-64 -ATATCATGGCTGACAGTAAAGTAATTTAATTGCACTGTTGCTATGACAACATACTGCTC
456150_ENSDARG00000012833_ZEBRAFISH_1704_1767/1-64 TATATTACAGCTGACAATAAAGTAATTTAATTGCACAGTTACCATGACAACGGCGGGCTC
* ** * ******* ****** ** * *** ** *** * ******** ***
456150_ENSG00000053254_HUMAN_2206_2269/1-64 TCTT
456150_ENSMUSG00000033713_MOUSE_7446_7509/1-64 TCTT
456150_SINFRUG00000137365_FUGU_7988_8049/1-64 ACT-
456150_ENSRNOG00000004709_RAT_7742_7803/1-64 TCT-
456150_ENSDARG00000012833_ZEBRAFISH_1704_1767/1-64 TGTT
*
456150-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 64
Mean pairwise identity: 83.39
Mean single sequence MFE: -13.66
Consensus MFE: -9.82
Energy contribution: -10.78
Covariance contribution: 0.96
Mean z-score: -2.24
Structure conservation index: 0.72
SVM decision value: 1.10
SVM RNA-class probability: 0.915486
Prediction: RNA
######################################################################
>456150_ENSG00000053254_HUMAN_2206_2269/1-64
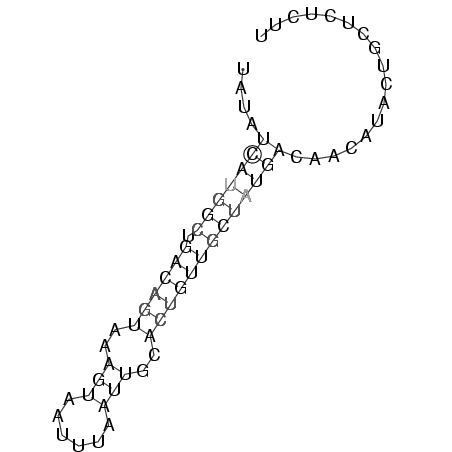
UAUAUCAUGGCUGACAGUAAAGUAAUUUAAUUGCACUGUUGCUAUGACAACAUGCUGCUCUCUU
....(((((((.((((((...(((((...))))))))))))))))))................. ( -15.50)
>456150_ENSMUSG00000033713_MOUSE_7446_7509/1-64
UAUAUCAUGGCUGACAGUAAAGUAAUUUAAUUGCACUGUUGCUAUGACAACAUACUGCUCUCUU
....(((((((.((((((...(((((...))))))))))))))))))................. ( -15.50)
>456150_SINFRUG00000137365_FUGU_7988_8049/1-64
ACAUUACGGCUGACAGUAAAGUCAUCUCAUUUUACUGUUGCUAUGACAACAAACUCCUCACU
...(((..((.(((((((((((......)))))))))))))..)))................ ( -14.20)
>456150_ENSRNOG00000004709_RAT_7742_7803/1-64
AUAUCAUGGCUGACAGUAAAGUAAUUUAAUUGCACUGUUGCUAUGACAACAUACUGCUCUCU
...(((((((.((((((...(((((...))))))))))))))))))................ ( -15.50)
>456150_ENSDARG00000012833_ZEBRAFISH_1704_1767/1-64
UAUAUUACAGCUGACAAUAAAGUAAUUUAAUUGCACAGUUACCAUGACAACGGCGGGCUCUGUU
........(((((.(((((((....))).))))..)))))........(((((......))))) ( -7.60)
>consensus
UAUAUCAUGGCUGACAGUAAAGUAAUUUAAUUGCACUGUUGCUAUGACAACAUACUGCUCUCUU
....(((((((.((((((..(((......)))..)))))))))))))................. ( -9.82 = -10.78 + 0.96)
456150-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004