Index >
Results for CNB 461739-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
461739_ENSMUSG00000034255_MOUSE_8785_8894/1-112 TACCAGCTCTGAGATGCCTTCGGCAATGGCTTTGTGCCAGGTGCTGATGATGGCTTCGGA
461739_SINFRUG00000131017_FUGU_9484_9593/1-112 -ACCAGTTGTCGTATGGTATCGGTCAGGACTCGGTGCCAGTCAACAATGATGCTTTCTGT
461739_ENSRNOG00000028569_RAT_8775_8884/1-112 TACCAGCTCCTCGATGCCTTCGGCAATGGCCTTGTGCCAGGTGCTGATGATGGCCTCTGA
461739_ENSDARG00000006344_ZEBRAFISH_6913_7022/1-112 -ACCAGCTGGTGTATGGTATCTAAGATAACTTTATGCCAGTCTTGGATGATGCTATCGGT
***** * *** ** * * ****** ****** ** *
461739_ENSMUSG00000034255_MOUSE_8785_8894/1-112 GTCGTGCTGGATCAGGTACTCC-GAGCCATCTCGGCTTCGAAGCTGGAGGG-
461739_SINFRUG00000131017_FUGU_9484_9593/1-112 GTCGTACTGTATCAGGAATTCCACCGCGTTCTTAGTTTTTAA-CTGGGGAGG
461739_ENSRNOG00000028569_RAT_8775_8884/1-112 GTCATGCTGGATCAGGTACTCT-GAGCCATCTCGGCTCCGGAGCTGGAGGG-
461739_ENSDARG00000006344_ZEBRAFISH_6913_7022/1-112 GTCGTACTGGATCAAGTATTCG-GCACCAGTCCGAGTCTTAAGCTGAATGAG
*** * *** **** * * ** * * * ***
461739-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 112
Mean pairwise identity: 65.37
Mean single sequence MFE: -44.60
Consensus MFE: -20.84
Energy contribution: -23.40
Covariance contribution: 2.56
Mean z-score: -2.78
Structure conservation index: 0.47
SVM decision value: 2.75
SVM RNA-class probability: 0.996817
Prediction: RNA
######################################################################
>461739_ENSMUSG00000034255_MOUSE_8785_8894/1-112
UACCAGCUCUGAGAUGCCUUCGGCAAUGGCUUUGUGCCAGGUGCUGAUGAUGGCUUCGGAGUCGUGCUGGAUCAGGUACUCCGAGCCAUCUCGGCUUCGAAGCUGGAGGG
..((((((.((((..(((.((((((.((((.....))))..)))))).(((((((.(((((((.((......)).).)))))))))))))..))))))).)))))).... ( -56.00)
>461739_SINFRUG00000131017_FUGU_9484_9593/1-112
ACCAGUUGUCGUAUGGUAUCGGUCAGGACUCGGUGCCAGUCAACAAUGAUGCUUUCUGUGUCGUACUGUAUCAGGAAUUCCACCGCGUUCUUAGUUUUUAACUGGGGAGG
.((.((((.....((((((((((....)).))))))))..)))).(((((((.....))))))).........))..........(.((((((((.....)))))))).) ( -30.50)
>461739_ENSRNOG00000028569_RAT_8775_8884/1-112
UACCAGCUCCUCGAUGCCUUCGGCAAUGGCCUUGUGCCAGGUGCUGAUGAUGGCCUCUGAGUCAUGCUGGAUCAGGUACUCUGAGCCAUCUCGGCUCCGGAGCUGGAGGG
..((((((((....((((...)))).((((.....))))((.(((((.((((((.((.((((...(((......))))))).))))))))))))).)))))))))).... ( -53.30)
>461739_ENSDARG00000006344_ZEBRAFISH_6913_7022/1-112
ACCAGCUGGUGUAUGGUAUCUAAGAUAACUUUAUGCCAGUCUUGGAUGAUGCUAUCGGUGUCGUACUGGAUCAAGUAUUCGGCACCAGUCCGAGUCUUAAGCUGAAUGAG
..(((((((((((.((((((...))).))).))))))((.((((((((((...)))(((((((((((......))))..))))))).))))))).))..)))))...... ( -38.60)
>consensus
_ACCAGCUCUUGGAUGCCAUCGGCAAUGACUUUGUGCCAGGCGCUGAUGAUGCCUUCGGAGUCGUACUGGAUCAGGUACUCC_GAGCCAUCUCGGCUCCGAAGCUGGAGGG_
..((((((.......((((((((((..(((.....)))...))))...)))))).(((((((((.(.(((.((.((....)).)).))).).)))))))))))))))..... (-20.84 = -23.40 + 2.56)
461739-rev.rnaz
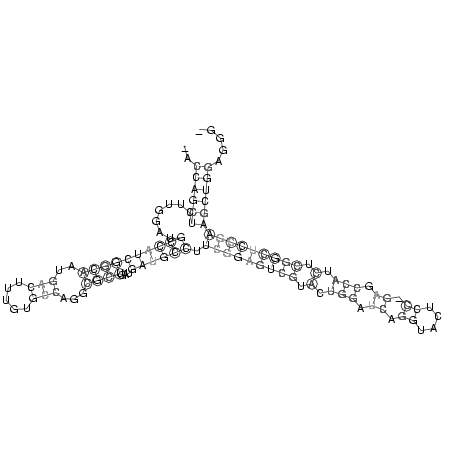
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004