Index >
Results for CNB 489516
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
489516_ENSG00000089213_HUMAN_5694_5848/1-156 -AGAATTGAAGAACTCACATGTGGTGGGATGGTCGAGCAGGTCCAGGAAGCCTTTGGCGA
489516_ENSRNOG00000001354_RAT_4711_4865/1-156 -AGAATTGAAGAGCTCACCTGTGGTGGGATGGTGGAGCAGGTCCAGGAAGCCTTTGGGGA
489516_ENSMUSG00000043679_MOUSE_5538_5692/1-156 -AGAATTGAAGAGCTCACCTGTGGTGGGATGGTGGAGCAGGTCCAGGAAGCCTTTGGTGA
489516_ENSDARG00000003846_ZEBRAFISH_6740_6894/1-156 AAGGGTGGAGGAGCTCACCTGTGGTGGGATGATGGAGCAGGTGCAGGAGGCGTTTGGAGA
** * ** ** ***** ************ * ******** ***** ** ***** **
489516_ENSG00000089213_HUMAN_5694_5848/1-156 GACCATGACCTCTGTTGTGTCTCTGTGTGCTCGTTACCCTATCGCTTGCGCAAATAGCAT
489516_ENSRNOG00000001354_RAT_4711_4865/1-156 GACCATGACGTCTGTTGTGTCTCTGTGTGCTCGGTACCCTATTGCTTGTGCGAACAGCAT
489516_ENSMUSG00000043679_MOUSE_5538_5692/1-156 GACCATGACCTCTGTCGTGTCTTTGTGTGCTCGGTACCCCATTGCTTGTGCGAACAGCAT
489516_ENSDARG00000003846_ZEBRAFISH_6740_6894/1-156 GACCATGACGTCAGTGGTGTCTCTGTGTGCCCGATACCCCATCGCCTGTGCCAACAGCAT
********* ** ** ****** ******* ** ***** ** ** ** ** ** *****
489516_ENSG00000089213_HUMAN_5694_5848/1-156 TGGACTCTTATGTACCATACCTTACACCAGGTAAGT
489516_ENSRNOG00000001354_RAT_4711_4865/1-156 TGGACTCCTGTGTACCATACCTTACACCAGGTGAGT
489516_ENSMUSG00000043679_MOUSE_5538_5692/1-156 TGGACTCCTGTGTACCATACCTTATACCAGGTGAGT
489516_ENSDARG00000003846_ZEBRAFISH_6740_6894/1-156 CGGCCTGCTCTGCACCATCCCCTACACCAGGCAAG-
** ** * ** ***** ** ** ****** **
//
489516.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 156
Mean pairwise identity: 86.50
Mean single sequence MFE: -57.50
Consensus MFE: -43.25
Energy contribution: -43.50
Covariance contribution: 0.25
Mean z-score: -1.41
Structure conservation index: 0.75
SVM decision value: -0.01
SVM RNA-class probability: 0.527445
Prediction: RNA
######################################################################
>489516_ENSG00000089213_HUMAN_5694_5848/1-156
AGAAUUGAAGAACUCACAUGUGGUGGGAUGGUCGAGCAGGUCCAGGAAGCCUUUGGCGAGACCAUGACCUCUGUUGUGUCUCUGUGUGCUCGUUACCCUAUCGCUUGCGCAAAUAGCAUUGGACUCUUAUGUACCAUACCUUACACCAGGUAAGU
...................((((((((.(((.((((((.(((.(((....))).)))(((((.(..((....))..))))))....))))))))).)))))))).(((.......))).(((((......)).)))(((((......)))))... ( -43.20)
>489516_ENSRNOG00000001354_RAT_4711_4865/1-156
AGAAUUGAAGAGCUCACCUGUGGUGGGAUGGUGGAGCAGGUCCAGGAAGCCUUUGGGGAGACCAUGACGUCUGUUGUGUCUCUGUGUGCUCGGUACCCUAUUGCUUGUGCGAACAGCAUUGGACUCCUGUGUACCAUACCUUACACCAGGUGAGU
...........(((((((((((..(((((((((..(((((((((..((((...(((((((((.(..((....))..))))))...((((...))))))))..))))((((.....))))))))..))))).)))))).)))..)).))))))))) ( -60.80)
>489516_ENSMUSG00000043679_MOUSE_5538_5692/1-156
AGAAUUGAAGAGCUCACCUGUGGUGGGAUGGUGGAGCAGGUCCAGGAAGCCUUUGGUGAGACCAUGACCUCUGUCGUGUCUUUGUGUGCUCGGUACCCCAUUGCUUGUGCGAACAGCAUUGGACUCCUGUGUACCAUACCUUAUACCAGGUGAGU
...........(((((((((..(((((((((((..(((((((((..((((...(((.(((((.(((((....))))))))))...((((...)))).)))..))))((((.....))))))))..))))).)))))).)).)))..))))))))) ( -57.30)
>489516_ENSDARG00000003846_ZEBRAFISH_6740_6894/1-156
AAGGGUGGAGGAGCUCACCUGUGGUGGGAUGAUGGAGCAGGUGCAGGAGGCGUUUGGAGAGACCAUGACGUCAGUGGUGUCUCUGUGUGCCCGAUACCCCAUCGCCUGUGCCAACAGCAUCGGCCUGCUCUGCACCAUCCCCUACACCAGGCAAG
...((((.(((..((((((...))))))..(((((.(((((.(((((.((((..((((((.(((((.......))))).))))))..)))))...........(((.((((.....)))).)))))))))))).))))).))).))))....... ( -68.70)
>consensus
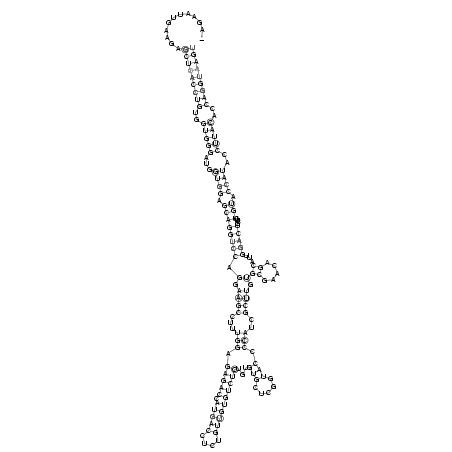
_AGAAUUGAAGAGCUCACCUGUGGUGGGAUGGUGGAGCAGGUCCAGGAAGCCUUUGGAGAGACCAUGACCUCUGUUGUGUCUCUGUGUGCUCGGUACCCCAUCGCUUGUGCGAACAGCAUUGGACUCCUGUGUACCAUACCUUACACCAGGUAAGU
............(((((((((..(((((...((((.((((((((.(.((((...(((.(((((.(((((....))))))))))...(((.....))).)))..)))).)((.....))...)))))....))).))))..)))))..))))))))) (-43.25 = -43.50 + 0.25)
489516.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004