Index >
Results for CNB 501416_6-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 GTCAAGC--TACTACCCTCGCTAACAATGCTCTGGCTTCTGGACCTGGGGAGCCAGGGTT
501416_ENSRNOG00000011386_RAT_133_729/1-680 GTCAAGC--TACTACCCTCGCTAACAATGCTCTGGCTTCTGGACCTGGGGAGCCAGGGTT
501416_ENSG00000135540_HUMAN_9403_9999/1-680 GTCATGC--TGCTGCCAGCACTAACTGTGCTCTGGCTCCGGGAGCGGGGCAGCCAGGGCT
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 AGACCACCGAGCTCCCTGCACTGATGCTGCTCTGACTGCGAGGCCTTAAGAGCCAGGGGT
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 ACACCAC--TACTACTTGCACTGATGCTGCTCTGGCTCCTTGGACGAAGCATCCAGGGGT
. .. .* :.** * *.**.* *******.** * *. * .. * ****** *
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 CCTCAAAGTCACTGCAGGGGCTCTGGGAAGGGGAGCTGCCG------CCCTTGCCAGG--
501416_ENSRNOG00000011386_RAT_133_729/1-680 CCTCACAGTCACTGCAGGGGCTCTGGGAAGGGGAACCGCTG------CCTTGGCCAGG--
501416_ENSG00000135540_HUMAN_9403_9999/1-680 CTTCCAAGTCACTGCAGGGGCTCTGGGAGGGCGAGCTGCCA------CTCTTGCCTGG--
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 CATCATAGTCGCTGCTGGGGCTGTGAGACGGCGAGGAAGGCGATGCCCCTTTTCCTTTCC
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 CCTCATAGTCACTACAAGGGCTATGAGATGGTGACGTGGAGGAT---CCTTTGCCTTT--
* **. ****.**.*:.***** **.** ** ** . * * **:
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 -------GAGGCTTAGCTGCAGTGAATGCTGGAGCGAGGCGATCAGGCTCTCATT-----
501416_ENSRNOG00000011386_RAT_133_729/1-680 -------GAGGCTTAGCTGCAGAGAATGCTGGAGTGAGGCGATCAGGCTCTCGTT-----
501416_ENSG00000135540_HUMAN_9403_9999/1-680 -------GAGGCTCAGCTGCAGCGAGTGCTGGAGTGTGGCGATCAGGCTCTCGTT-----
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 CTCCCCCCAGACCCATCTGTAGACTCTGCTGCAGGCTGGCAATGAGCGTCTCACTGAGCA
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 -------AAGCCCATTCTGAAGTGACTGTTGGAGAGTGGCAATCAATGATTCATTGAGAA
** * : *** ** : ** ** ** :***.** *. : **. *
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 GAGCAC--------------------CTGACC-GT--TT-GT-CTG-------GTTGTTC
501416_ENSRNOG00000011386_RAT_133_729/1-680 GAGCAC--------------------CTGCCC-AT--TT-GT-CTG-------GTTGTTC
501416_ENSG00000135540_HUMAN_9403_9999/1-680 GAGCAC--------------------CTGCCC-GT--TG-CA-CTG-------GCTGCTC
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 TGGCTCCATTTGCATCGTTAGCTCCATTCACC-GTGCTGACACCTCCGACGGGCTTTTTC
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 GAGAACCATTCCTAATATTTG----ACTGCCCAAT-ATT-GG-CAGAGGGAGAGCTGTTT
.*.:* * .** .* * *: * *
501416_ENSMUSG00000039825_MOUSE_133_713/1-680 TTTTTGGGAATCCTTCGCAGAGAATCGGTCCGGGATGGTGGCAGGGGTGGTTTCTTTGCT
501416_ENSRNOG00000011386_RAT_133_729/1-680 TTCTTGGGAATCCTTCGCAGAGAATCTGTCCGGGATGGTGGCGGTGGTGGTTTTTTTGCT
501416_ENSG00000135540_HUMAN_9403_9999/1-680 TTCTTGGGAATCCTGCGGAGGGAGTCTGTCCGGGAGGGTGGCAGGGGAGGCTTCTTTGCT
501416_SINFRUG00000136522_FUGU_3948_4614/1-680 GGGCCTGGCTTGCGTCTGAGAGAGTCTGTTCGAGCCGGAGGCAGCGGTGGCTTCTTAGAC
501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680 TTCCCAGGCTTTCTCCGTAGAGAGTCTGTGCGTGCAGGTGGAAGGGGTGGTTTCTTAGGC
**.:* * * **.**.** ** ** *. **:**..* **:** ** **:*
501416_6-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 62.99
Mean single sequence MFE: -114.96
Consensus MFE: -44.57
Energy contribution: -46.17
Covariance contribution: 1.60
Mean z-score: -2.42
Structure conservation index: 0.39
SVM decision value: 1.50
SVM RNA-class probability: 0.960532
Prediction: RNA
######################################################################
>501416_ENSMUSG00000039825_MOUSE_133_713/1-680
GUCAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGGGAGCUGCCGCCCUUGCCAGGGAGGCUUAGCUGCAGUGAAUGCUGGAGCGAGGCGAUCAGGCUCUCAUUGAGCACCUGACCGUUUGUCUGGUUGUUCUUUUUGGGAAUCCUUCGCAGAGAAUCGGUCCGGGAUGGUGGCAGGGGUGGUUUCUUUGCU
((.(((..(((((((..((((.((..((....))(((((((((((((((((....))))))))...((.((((((((..(((.(((((((((((((((.((((....)))).)))...(((.((((....)))).)))..........)))))))...(((((((.(((.....))).)).)))))))))).)))..)))).)))).))...))))))))))).))))..)))))))..))).)). ( -111.90)
>501416_ENSRNOG00000011386_RAT_133_729/1-680
GUCAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCACAGUCACUGCAGGGGCUCUGGGAAGGGGAACCGCUGCCUUGGCCAGGGAGGCUUAGCUGCAGAGAAUGCUGGAGUGAGGCGAUCAGGCUCUCGUUGAGCACCUGCCCAUUUGUCUGGUUGUUCUUCUUGGGAAUCCUUCGCAGAGAAUCUGUCCGGGAUGGUGGCGGUGGUGGUUUUUUUGCU
...(((((((((((.((((((.....((((((((((((.......))))))))))))((((.((((((.((((..(((.((..(((((((.(((((......(((.((((((.((((.(((.(((......))).))).))))..))..((((.....)))).)))).)))......))))).)))))))..))...)))..)))).))..))))..)))))))))).)))))))))))....... ( -106.20)
>501416_ENSG00000135540_HUMAN_9403_9999/1-680
GUCAUGCUGCUGCCAGCACUAACUGUGCUCUGGCUCCGGGAGCGGGGCAGCCAGGGCUCUUCCAAGUCACUGCAGGGGCUCUGGGAGGGCGAGCUGCCACUCUUGCCUGGGAGGCUCAGCUGCAGCGAGUGCUGGAGUGUGGCGAUCAGGCUCUCGUUGAGCACCUGCCCGUUGCACUGGCUGCUCUUCUUGGGAAUCCUGCGGAGGGAGUCUGUCCGGGAGGGUGGCAGGGGAGGCUUCUUUGCU
......(((..((((((((.....))))..))))..))).(((((((.((((...(((((((((((((........)))).)))))))))...((((((((((..((((((((((((..((((((.(((((((..((((..(((..(((((((.....))))..)))..)))..))))))).))))(((....)))..))))))...)))))).))))))))))))))))....)))).))))))) ( -120.50)
>501416_SINFRUG00000136522_FUGU_3948_4614/1-680
AGACCACCGAGCUCCCUGCACUGAUGCUGCUCUGACUGCGAGGCCUUAAGAGCCAGGGGUCAUCAUAGUCGCUGCUGGGGCUGUGAGACGGCGAGGAAGGCGAUGCCCCUUUUCCUUUCCCUCCCCCCAGACCCAUCUGUAGACUCUGCUGCAGGCUGGCAAUGAGCGUCUCACUGAGCAUGGCUCCAUUUGCAUCGUUAGCUCCAUUCACCGUGCUGACACCUCCGACGGGCUUUUUCGGGCCUGGCUUGCGUCUGAGAGAGUCUGUUCGAGCCGGAGGCAGCGGUGGCUUCUUAGAC
((.((((((.((..((.((...(((((.((((.....(.(((((((((...((((((((((.((((((((.(....).))))))))((.((.(((((((((...)))....)))))).)).))......))))).(((((((......))))))))))))..)))).))))).).))))(((....)))..)))))((((((.(........).))))))...((.((((((((((((((((((......).))))))))))))))))).)))).))..))..))))))))........ ( -126.00)
>501416_ENSDARG00000026402_ZEBRAFISH_375_1001/1-680
ACACCACUACUACUUGCACUGAUGCUGCUCUGGCUCCUUGGACGAAGCAUCCAGGGGUCCUCAUAGUCACUACAAGGGCUAUGAGAUGGUGACGUGGAGGAUCCUUUGCCUUUAAGCCCAUUCUGAAGUGACUGUUGGAGAGUGGCAAUCAAUGAUUCAUUGAGAAGAGAACCAUUCCUAAUAUUUGACUGCCCAAUAUUGGCAGAGGGAGAGCUGUUUUUCCCAGGCUUUCUCCGUAGAGAGUCUGUGCGUGCAGGUGGAAGGGGUGGUUUCUUAGGC
.((((.((.((((((((((((((((..(((((((.(((((((.......))))))))))((.(((((((((.((.(((((..(((..(....)(..(((....)))..))))..)))))....)).))))))))).))))))..)).))))..(((((.(((.(.(((((.((...............(((((.......))))).(((((((....))))))).)).))))).).))).))))).....)))))))))).)).))))........... ( -110.20)
>consensus
GUCAAGC__UACUACCCGCACUAACAAUGCUCUGGCUCCUGGACCUGGGGAGCCAGGGGUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGCGAGCUGCCG______CCUUUGCCUGG_________GAGGCUCAGCUGCAGAGAAUGCUGGAGUGUGGCGAUCAGGCUCUCAUU_____GAGCAC____________________CUGCCC_GU__UU_GA_CUG_______GUUGUUCUUCUUGGGAAUCCUUCGCAGAGAGUCUGUCCGGGAUGGUGGCAGGGGUGGUUUCUUUGCU
..........((((((............(((((((((((((....)))).)))))))))........(((((((((((..(((((.((((((((((.((...............................((((.(((.(((......))).))).)))).........(((........)))..................................................)).)))))..........))))).)))))..))))).......))))))...))))))......... (-44.57 = -46.17 + 1.60)
501416_6-rev.rnaz
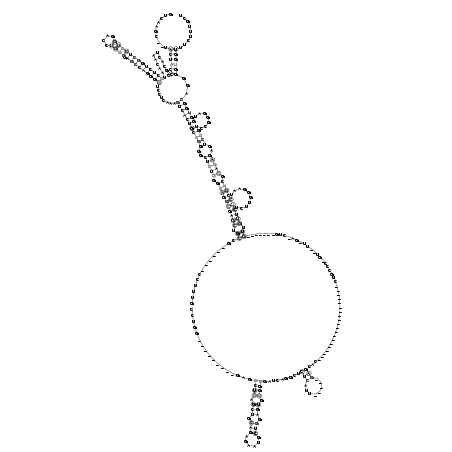
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004