Index >
Results for CNB 501429_8-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
501429_ENSRNOG00000011386_RAT_133_730/1-682 -TGCTTGTGTCGCTCTGAGATGGAGTGAGCCCGCACAGAGAATACACGT-TGGGAGT--G
501429_ENSMUSG00000039825_MOUSE_133_716/1-682 CTACTTGTGTCGCTCTGGGACGGAGTGACCCCACACAGAGAATACACGT-TGGGAGT--G
501429_ENSDARG00000026402_ZEBRAFISH_375_1002/1-682 -TACTTGTGTCACTATGAGATGGTGTGACGGGACAGATGGAGTATACATGAGCCATTCCT
501429_SINFRUG00000136522_FUGU_3948_4615/1-682 -TGCTGGTCTCACTGTGAGTGGGGGTCACGTGGCACAGAGAGTAAACATTAGAGACACCA
501429_ENSG00000135540_HUMAN_9403_10000/1-682 -TGCTTGTGTCACTCTGCGATGGCGTGGCCCCGCACAGGGAGTAGACAT-TGGGGGT--G
*.** ** **.** ** *: ** ** . .** * .**.** **.* :* . :
501429_ENSRNOG00000011386_RAT_133_730/1-682 G---------TGGTG--GAG--GTCAAGC--TACTACCCTCGCTAACAATGCTCTGGCTT
501429_ENSMUSG00000039825_MOUSE_133_716/1-682 G---------TGGTG--GAG--GTCAAGC--TACTACCCTCGCTAACAATGCTCTGGCTT
501429_ENSDARG00000026402_ZEBRAFISH_375_1002/1-682 G---------TAGCT--GAT--ACACCAC--TACTACTTGCACTGATGCTGCTCTGGCTC
501429_SINFRUG00000136522_FUGU_3948_4615/1-682 GCGCAAGCATTAGCTCCTATCGAGACCACCGAGCTCCCTGCACTGATGCTGCTCTGACTG
501429_ENSG00000135540_HUMAN_9403_10000/1-682 G---------TGGCG--GAA--GTCATGC--TGCTGCCAGCACTAACTGTGCTCTGGCTC
* *.* * . .. .* :.** * *.**.* *******.**
501429_ENSRNOG00000011386_RAT_133_730/1-682 CTGGACCTGGGGAGCCAGGGTTCCTCACAGTCACTGCAGGGGCTCTGGGAAGGGGAACCG
501429_ENSMUSG00000039825_MOUSE_133_716/1-682 CTGGACCTGGGGAGCCAGGGTTCCTCAAAGTCACTGCAGGGGCTCTGGGAAGGGGAGCTG
501429_ENSDARG00000026402_ZEBRAFISH_375_1002/1-682 CTTGGACGAAGCATCCAGGGGTCCTCATAGTCACTACAAGGGCTATGAGATGGTGACGTG
501429_SINFRUG00000136522_FUGU_3948_4615/1-682 CGAGGCCTTAAGAGCCAGGGGTCATCATAGTCGCTGCTGGGGCTGTGAGACGGCGAGGAA
501429_ENSG00000135540_HUMAN_9403_10000/1-682 CGGGAGCGGGGCAGCCAGGGCTCTTCCAAGTCACTGCAGGGGCTCTGGGAGGGCGAGCTG
* *. * .. * ****** ** **. ****.**.*:.***** **.** ** ** .
501429_ENSRNOG00000011386_RAT_133_730/1-682 CTG------CCTTGGCCAGG---------GAGGCTTAGCTGCAGAGAATGCTGGAGTGAG
501429_ENSMUSG00000039825_MOUSE_133_716/1-682 CCG------CCCTTGCCAGG---------GAGGCTTAGCTGCAGTGAATGCTGGAGCGAG
501429_ENSDARG00000026402_ZEBRAFISH_375_1002/1-682 GAGGAT---CCTTTGCCTTT---------AAGCCCATTCTGAAGTGACTGTTGGAGAGTG
501429_SINFRUG00000136522_FUGU_3948_4615/1-682 GGCGATGCCCCTTTTCCTTTCCCTCCCCCCAGACCCATCTGTAGACTCTGCTGCAGGCTG
501429_ENSG00000135540_HUMAN_9403_10000/1-682 CCA------CTCTTGCCTGG---------GAGGCTCAGCTGCAGCGAGTGCTGGAGTGTG
* * **: ** * : *** ** : ** ** ** :*
501429_ENSRNOG00000011386_RAT_133_730/1-682 GCGATCAGGCTCTCGTT-----GAGCAC--------------
501429_ENSMUSG00000039825_MOUSE_133_716/1-682 GCGATCAGGCTCTCATT-----GAGCAC--------------
501429_ENSDARG00000026402_ZEBRAFISH_375_1002/1-682 GCAATCAATGATTCATTGAGAAGAGAACCATTCCTAATATTT
501429_SINFRUG00000136522_FUGU_3948_4615/1-682 GCAATGAGCGTCTCACTGAGCATGGCTCCATTTGCATCGTTA
501429_ENSG00000135540_HUMAN_9403_10000/1-682 GCGATCAGGCTCTCGTT-----GAGCAC--------------
**.** *. : **. * .*.:*
501429_8-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 282
Mean pairwise identity: 62.86
Mean single sequence MFE: -101.64
Consensus MFE: -39.96
Energy contribution: -39.92
Covariance contribution: -0.04
Mean z-score: -1.81
Structure conservation index: 0.39
SVM decision value: 0.11
SVM RNA-class probability: 0.586558
Prediction: RNA
######################################################################
>501429_ENSRNOG00000011386_RAT_133_730/1-682
UGCUUGUGUCGCUCUGAGAUGGAGUGAGCCCGCACAGAGAAUACACGUUGGGAGUGGUGGUGGAGGUCAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCACAGUCACUGCAGGGGCUCUGGGAAGGGGAACCGCUGCCUUGGCCAGGGAGGCUUAGCUGCAGAGAAUGCUGGAGUGAGGCGAUCAGGCUCUCGUUGAGCAC
((.((((.(((((((.((.((.((((((((.((.(((((.......((((((((.((((((((........))))))))))).)))))....)))))))((((((.((.(((.(((((((((((((..(((...))).))))))))))....((....))))).))).)))))))).)))))).)).)).......))))))))).)))).)).((((.....)))).. ( -100.20)
>501429_ENSMUSG00000039825_MOUSE_133_716/1-682
CUACUUGUGUCGCUCUGGGACGGAGUGACCCCACACAGAGAAUACACGUUGGGAGUGGUGGUGGAGGUCAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGGGAGCUGCCGCCCUUGCCAGGGAGGCUUAGCUGCAGUGAAUGCUGGAGCGAGGCGAUCAGGCUCUCAUUGAGCAC
((..((((.(((((((((..(((((((....))).(((((.......((((((((.((((((((........))))))))))).)))))....)))))...))))..))((((((((....))))))))...(((((((((.((((((.......))))))(((.((((....)))).)))....))))))))).....))))))).))))..))((((.....)))).. ( -106.90)
>501429_ENSDARG00000026402_ZEBRAFISH_375_1002/1-682
UACUUGUGUCACUAUGAGAUGGUGUGACGGGACAGAUGGAGUAUACAUGAGCCAUUCCUGUAGCUGAUACACCACUACUACUUGCACUGAUGCUGCUCUGGCUCCUUGGACGAAGCAUCCAGGGGUCCUCAUAGUCACUACAAGGGCUAUGAGAUGGUGACGUGGAGGAUCCUUUGCCUUUAAGCCCAUUCUGAAGUGACUGUUGGAGAGUGGCAAUCAAUGAUUCAUUGAGAAGAGAACCAUUCCUAAUAUUU
..(((.(.(((..(((((.(((((((.(((.((((((((..((....))..))))..))))..))).)))))))..........((.((((((..(((((((.(((((((.......))))))))))((.(((((((((.((.(((((..(((..(....)(..(((....)))..))))..)))))....)).))))))))).))))))..)).)))).)).)))))))).).)))................. ( -85.60)
>501429_SINFRUG00000136522_FUGU_3948_4615/1-682
UGCUGGUCUCACUGUGAGUGGGGGUCACGUGGCACAGAGAGUAAACAUUAGAGACACCAGCGCAAGCAUUAGCUCCUAUCGAGACCACCGAGCUCCCUGCACUGAUGCUGCUCUGACUGCGAGGCCUUAAGAGCCAGGGGUCAUCAUAGUCGCUGCUGGGGCUGUGAGACGGCGAGGAAGGCGAUGCCCCUUUUCCUUUCCCUCCCCCCAGACCCAUCUGUAGACUCUGCUGCAGGCUGGCAAUGAGCGUCUCACUGAGCAUGGCUCCAUUUGCAUCGUUA
((((((((((.(((((.(((.....)))....))))).((((........(......).((....))....)))).....)))))))..(((((....(((....)))(((((.....(.(((((((((...((((((((((.((((((((.(....).))))))))((.((.(((((((((...)))....)))))).)).))......))))).(((((((......))))))))))))..)))).))))).).))))).))))).....)))...... ( -103.40)
>501429_ENSG00000135540_HUMAN_9403_10000/1-682
UGCUUGUGUCACUCUGCGAUGGCGUGGCCCCGCACAGGGAGUAGACAUUGGGGGUGGUGGCGGAAGUCAUGCUGCUGCCAGCACUAACUGUGCUCUGGCUCCGGGAGCGGGGCAGCCAGGGCUCUUCCAAGUCACUGCAGGGGCUCUGGGAGGGCGAGCUGCCACUCUUGCCUGGGAGGCUCAGCUGCAGCGAGUGCUGGAGUGUGGCGAUCAGGCUCUCGUUGAGCAC
...........(((((((.((((...(((((((.(..(((((((((((.(..((((.((((((.((.....)).)))))).))))..).))).))).)))))..).))))))).)))).((((......))))..)))))))((((.(.((((((((..((((((....(((.....)))...(((.((((....)))).))))))))).))..)))))).).)))).. ( -112.10)
>consensus
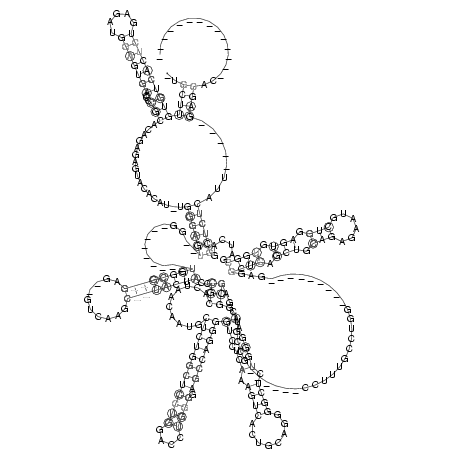
_UGCUUGUGUCACUCUGAGAUGGAGUGACCCCGCACAGAGAGUACACAU_UGGGAGU__GG_________UGGCG__GAG__GUCAAGC__UACUACCCGCACUAACAAUGCUCUGGCUCCUGGACCUGGGGAGCCAGGGGUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGCGAGCUGCCG______CCUUUGCCUGG_________GAGGCUCAGCUGCAGAGAAUGCUGGAGUGUGGCGAUCAGGCUCUCAUU_____GAGCAC______________
..((((((((((((((.....)))))))...))).................((((((.............((((((((..........))))))))..(((..........((((((((((((....)))).))))))))(((.(((.((((........)))).)))))).))).....................................((((.(((.(((......))).))).))))......))))))........))))................ (-39.96 = -39.92 + -0.04)
501429_8-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004