Index >
Results for CNB 70843_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
70843_ENSDARG00000022900_ZEBRAFISH_290_655/1-368 -TCACTTGGAGCTGGTGTATTTGGTCACGGCCTTTGTTCCCTCAGACACTGCGTGTTTGG
70843_SINFRUG00000141037_FUGU_314_680/1-368 -CTATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGG
70843_ENSG00000124518_HUMAN_318_684/1-368 -TTACTTGGAGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCCGACACCGCGTGCTTGG
70843_ENSRNOG00000021192_RAT_336_703/1-368 CTCACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACAGCGTGCTTGG
70843_ENSMUSG00000049245_MOUSE_296_663/1-368 CTCACTTGGAGCTGGTGTACTTAGTGACAGCCTTGGTGCCCTCCGACACGGCGTGCTTGG
70843_ENSDARG00000022900_ZEBRAFISH_290_655/1-368 CCAGTTCTCCGGGCAGGAGCAGGCGCACGGCTGTCTGGATCTCCCGGGATGTGATAGTGG
70843_SINFRUG00000141037_FUGU_314_680/1-368 CCAGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGG
70843_ENSG00000124518_HUMAN_318_684/1-368 CCAGCTCCCCCGGAAGCAGCAGGCGCACGGCGGTCTGGATCTCCCTGGAGGTGATGGTCG
70843_ENSRNOG00000021192_RAT_336_703/1-368 CCAACTCCCCGGGAAGCAACAGACGCACCGCCGTCTGGATCTCCCGGGACGTGATGGTCG
70843_ENSMUSG00000049245_MOUSE_296_663/1-368 CCAGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCG
70843_ENSDARG00000022900_ZEBRAFISH_290_655/1-368 AGCGCTTGTTGTAATGCGCCAGGCGCGACGCTTCTCCGGCGATGCGCTCGAAGATGTCGT
70843_SINFRUG00000141037_FUGU_314_680/1-368 CGCGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGT
70843_ENSG00000124518_HUMAN_318_684/1-368 AGCGCTTGTTGTAGTGCGCCAGGCGGGAAGCCTCGCTTGCGATGCGCTCGAAGATGTCGT
70843_ENSRNOG00000021192_RAT_336_703/1-368 AGCGCTTGTTGTAATGAGCCAGGCGCGACGCCTCGCCCGCGATGCGCTCGAAGATGTCGT
70843_ENSMUSG00000049245_MOUSE_296_663/1-368 AGCGCTTGTTGTAATGCGCCAGGCGGGACGCCTCGCCCGCGATGCGCTCGAAGATGTCGT
70843_ENSDARG00000022900_ZEBRAFISH_290_655/1-368 TTACAAATGAGTTCATAATGCCCATCGCCTTTGAGGAGATGCCAGTGTCCGGGTGGACTT
70843_SINFRUG00000141037_FUGU_314_680/1-368 TCACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCT
70843_ENSG00000124518_HUMAN_318_684/1-368 TGACGAAGGAGTTCATGATTCCCATGGCCTTAGAAGAGATGCCGGTGTCGGGGTGGACCT
70843_ENSRNOG00000021192_RAT_336_703/1-368 TCACGAACGAGTTCATTATGCCCATGGCCTTGGAAGAGATGCCCGTGTCCGGATGCACTT
70843_ENSMUSG00000049245_MOUSE_296_663/1-368 TCACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTT
70843_ENSDARG00000022900_ZEBRAFISH_290_655/1-368 GTTTTAGTACTTTGTACACGTAAATGGCGTAACTCTCTTTCCTGGTCTTACGCCTTTTCT
70843_SINFRUG00000141037_FUGU_314_680/1-368 GCTTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCT
70843_ENSG00000124518_HUMAN_318_684/1-368 GCTTCAGCACCTTGTACACGTACACGGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCT
70843_ENSRNOG00000021192_RAT_336_703/1-368 GCTTAAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCT
70843_ENSMUSG00000049245_MOUSE_296_663/1-368 GCTTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCT
70843_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 86.02
Mean single sequence MFE: -128.61
Consensus MFE: -96.94
Energy contribution: -99.74
Covariance contribution: 2.80
Mean z-score: -1.61
Structure conservation index: 0.75
SVM decision value: 0.13
SVM RNA-class probability: 0.596432
Prediction: RNA
######################################################################
>70843_ENSDARG00000022900_ZEBRAFISH_290_655/1-368
UCACUUGGAGCUGGUGUAUUUGGUCACGGCCUUUGUUCCCUCAGACACUGCGUGUUUGGCCAGUUCUCCGGGCAGGAGCAGGCGCACGGCUGUCUGGAUCUCCCGGGAUGUGAUAGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCUUCUCCGGCGAUGCGCUCGAAGAUGUCGUUUACAAAUGAGUUCAUAAUGCCCAUCGCCUUUGAGGAGAUGCCAGUGUCCGGGUGGACUUGUUUUAGUACUUUGUACACGUAAAUGGCGUAACUCUCUUUCCUGGUCUUACGCCUUUUCU
......((((...((((((..(((.((.......((((.(((.(((((((((((((((((((...((((.....))))(((((((.(.((((((.(.((((....)))).))))))).).)))))))......)).))))))))))..((.((((((((((((.((.....((..(((((....)))))..)).....)).))))))).....))))).))))))))).))).))))........)))))..))))))......(((((((..((.......))..)))))))..)))) ( -113.16)
>70843_SINFRUG00000141037_FUGU_314_680/1-368
CUAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCU
......(((((..((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))......(((((((..(........)...)))))))))))). ( -125.80)
>70843_ENSG00000124518_HUMAN_318_684/1-368
UUACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCCGGAAGCAGCAGGCGCACGGCGGUCUGGAUCUCCCUGGAGGUGAUGGUCGAGCGCUUGUUGUAGUGCGCCAGGCGGGAAGCCUCGCUUGCGAUGCGCUCGAAGAUGUCGUUGACGAAGGAGUUCAUGAUUCCCAUGGCCUUAGAAGAGAUGCCGGUGUCGGGGUGGACCUGCUUCAGCACCUUGUACACGUACACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCU
......(((((((((((((.(((((.(((....((...)).))).))))).))))...)))))))))...((((((.((((((((((.((((..((((((((....(((((.((((((((((((..(((((((.(((..((((.....)))))))))))))))))))))).((.(((((.(((......))).))))))))))).)))))....))))).)))(((((((((((...(((...)))))))))).))))......((((.....)))))))).).))))))))))))))) ( -137.50)
>70843_ENSRNOG00000021192_RAT_336_703/1-368
CUCACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACAGCGUGCUUGGCCAACUCCCCGGGAAGCAACAGACGCACCGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGAGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUUAUGCCCAUGGCCUUGGAAGAGAUGCCCGUGUCCGGAUGCACUUGCUUAAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCU
.......(((((..((((((..((((...((...(((((..((((((((.((((.(((..((((...(((((((.....((((((......))))))....)))))))..((.((((((((((((..(((((...(.((.(((((...))))).))).))))))))))))).((..(((((....)))))..)).......)))).)).))))..)))))))..))))))))..))))).)).....))))..))))))(((......((((((........))))))...)))))))). ( -127.00)
>70843_ENSMUSG00000049245_MOUSE_296_663/1-368
CUCACUUGGAGCUGGUGUACUUAGUGACAGCCUUGGUGCCCUCCGACACGGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCU
.......(((((((((((((...(((((((....(((((.((((((((((((((.(((..((((..((..((((((((((((((((.((((((((((..((....))..)).)))))))).)))))))))))...((((...(((((.((...)))))))...)))).....((..(((((....)))))..))....)))))..))..))))..)))))))).))))))))).)))))(((....)))..)))).))))))))))).((((((........))))))......))))). ( -139.60)
>consensus
_UCACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACGGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCCGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUACACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCU
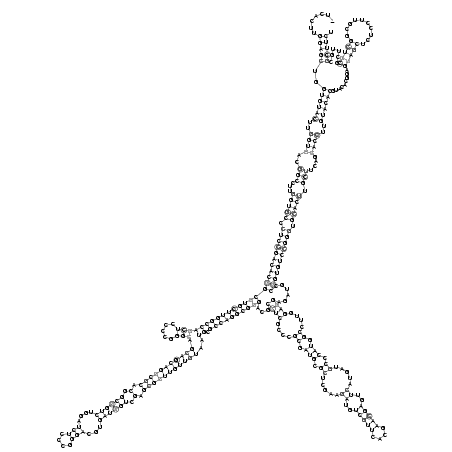
.......(((((..((((((..((((..(((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.(.((((((....(((....)))...)))))).).)))))))))))..)).))))))))))...((((....((.(((.((.....((..(((((....)))))..)).....)).))).))....))))......))))))))))).))))).)))....))))..)))))).........(((((..........)))))......))))). (-96.94 = -99.74 + 2.80)
70843_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004