Index >
Results for CNB 72961_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
72961_SINFRUG00000141037_FUGU_314_691/1-379 CTATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGGC
72961_ENSMUSG00000049150_MOUSE_327_703/1-379 -CACTTGGAGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCCGACACCGCGTGCTTGGC
72961_ENSG00000183868_HUMAN_301_677/1-379 -TACTTAGCGCTGGTGTACTTGGTGACGGCCTTAGTACCCTCGGACACGGCGTGCTTGGC
72961_ENSDARG00000022900_ZEBRAFISH_291_667/1-379 -CACTTGGAGCTGGTGTATTTGGTCACGGCCTTTGTTCCCTCAGACACTGCGTGTTTGGC
72961_ENSRNOG00000021192_RAT_338_714/1-379 -CACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACAGCGTGCTTGGC
72961_SINFRUG00000141037_FUGU_314_691/1-379 CAGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGGC
72961_ENSMUSG00000049150_MOUSE_327_703/1-379 CAGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCGA
72961_ENSG00000183868_HUMAN_301_677/1-379 CAACTCCCCAGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCTGGAGGTGATGGTCGA
72961_ENSDARG00000022900_ZEBRAFISH_291_667/1-379 CAGTTCTCCGGGCAGGAGCAGGCGCACGGCTGTCTGGATCTCCCGGGATGTGATAGTGGA
72961_ENSRNOG00000021192_RAT_338_714/1-379 CAACTCCCCGGGAAGCAACAGACGCACCGCCGTCTGGATCTCCCGGGACGTGATGGTCGA
72961_SINFRUG00000141037_FUGU_314_691/1-379 GCGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGTT
72961_ENSMUSG00000049150_MOUSE_327_703/1-379 GCGCTTGTTGTAATGCGCCAGGCGGGACGCCTCGCTCGCGATGCGCTCGAAGATGTCGTT
72961_ENSG00000183868_HUMAN_301_677/1-379 GCGCTTGTTGTAATGCGCCAGGCGGGAAGCCTCACCTGCGATGCGCTCGAAAATGTCGTT
72961_ENSDARG00000022900_ZEBRAFISH_291_667/1-379 GCGCTTGTTGTAATGCGCCAGGCGCGACGCTTCTCCGGCGATGCGCTCGAAGATGTCGTT
72961_ENSRNOG00000021192_RAT_338_714/1-379 GCGCTTGTTGTAATGAGCCAGGCGCGACGCCTCGCCCGCGATGCGCTCGAAGATGTCGTT
72961_SINFRUG00000141037_FUGU_314_691/1-379 CACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCTG
72961_ENSMUSG00000049150_MOUSE_327_703/1-379 CACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTTG
72961_ENSG00000183868_HUMAN_301_677/1-379 CACAAACGAATTCATGATGCCCATGGCCTTGGACGAAATGCCGGTGTCAGGGTGGACCTG
72961_ENSDARG00000022900_ZEBRAFISH_291_667/1-379 TACAAATGAGTTCATAATGCCCATCGCCTTTGAGGAGATGCCAGTGTCCGGGTGGACTTG
72961_ENSRNOG00000021192_RAT_338_714/1-379 CACGAACGAGTTCATTATGCCCATGGCCTTGGAAGAGATGCCCGTGTCCGGATGCACTTG
72961_SINFRUG00000141037_FUGU_314_691/1-379 CTTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCTT
72961_ENSMUSG00000049150_MOUSE_327_703/1-379 CTTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTT
72961_ENSG00000183868_HUMAN_301_677/1-379 CTTCAGAACCTTGTACACATAGATGGAATAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTT
72961_ENSDARG00000022900_ZEBRAFISH_291_667/1-379 TTTTAGTACTTTGTACACGTAAATGGCGTAACTCTCTTTCCTGGTCTTACGCCTTTTCTT
72961_ENSRNOG00000021192_RAT_338_714/1-379 CTTAAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTT
72961_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 85.60
Mean single sequence MFE: -125.87
Consensus MFE: -93.67
Energy contribution: -96.35
Covariance contribution: 2.68
Mean z-score: -1.57
Structure conservation index: 0.74
SVM decision value: 0.03
SVM RNA-class probability: 0.547665
Prediction: RNA
######################################################################
>72961_SINFRUG00000141037_FUGU_314_691/1-379
CUAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCUU
......(((((..((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))......(((((((..(........)...)))))))))))).. ( -125.80)
>72961_ENSMUSG00000049150_MOUSE_327_703/1-379
CACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGACGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
.....(((((..((((((..((((..(((...(((((.(((((((((((....)..(((...((((((((((((((((((((((.((((((((((..((....))..)).)))))))).)))))))))))..)))((((((((((.(((.(((....))).))))))...((..(((((....)))))..)).....)))..)))).)))).))))..))))))))))))).))))).)))....))))..))))))(((......((((((........))))))...)))))))).. ( -141.30)
>72961_ENSG00000183868_HUMAN_301_677/1-379
UACUUAGCGCUGGUGUACUUGGUGACGGCCUUAGUACCCUCGGACACGGCGUGCUUGGCCAACUCCCCAGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCUGGAGGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGAAGCCUCACCUGCGAUGCGCUCGAAAAUGUCGUUCACAAACGAAUUCAUGAUGCCCAUGGCCUUGGACGAAAUGCCGGUGUCAGGGUGGACCUGCUUCAGAACCUUGUACACAUAGAUGGAAUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
......((.(((((((((((((.((.((((..(((((((........)).))))).))))...)).)))))..(((((((((((.((((((((...(((((....))))))))))))).)))))))))))..))))))))))(((((((.......((((.((((.........((((((((....)...(((((.....)))))...)))))))...((((((((((((((.((......))...)))))).))))......(((.......)))...)))).))))))))))))))) ( -122.01)
>72961_ENSDARG00000022900_ZEBRAFISH_291_667/1-379
CACUUGGAGCUGGUGUAUUUGGUCACGGCCUUUGUUCCCUCAGACACUGCGUGUUUGGCCAGUUCUCCGGGCAGGAGCAGGCGCACGGCUGUCUGGAUCUCCCGGGAUGUGAUAGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCUUCUCCGGCGAUGCGCUCGAAGAUGUCGUUUACAAAUGAGUUCAUAAUGCCCAUCGCCUUUGAGGAGAUGCCAGUGUCCGGGUGGACUUGUUUUAGUACUUUGUACACGUAAAUGGCGUAACUCUCUUUCCUGGUCUUACGCCUUUUCUU
.....((((...((((((..(((.((.......((((.(((.(((((((((((((((((((...((((.....))))(((((((.(.((((((.(.((((....)))).))))))).).)))))))......)).))))))))))..((.((((((((((((.((.....((..(((((....)))))..)).....)).))))))).....))))).))))))))).))).))))........)))))..))))))......(((((((..((.......))..)))))))..)))). ( -113.26)
>72961_ENSRNOG00000021192_RAT_338_714/1-379
CACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACAGCGUGCUUGGCCAACUCCCCGGGAAGCAACAGACGCACCGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGAGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUUAUGCCCAUGGCCUUGGAAGAGAUGCCCGUGUCCGGAUGCACUUGCUUAAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
.....(((((..((((((..((((...((...(((((..((((((((.((((.(((..((((...(((((((.....((((((......))))))....)))))))..((.((((((((((((..(((((...(.((.(((((...))))).))).))))))))))))).((..(((((....)))))..)).......)))).)).))))..)))))))..))))))))..))))).)).....))))..))))))(((......((((((........))))))...)))))))).. ( -127.00)
>consensus
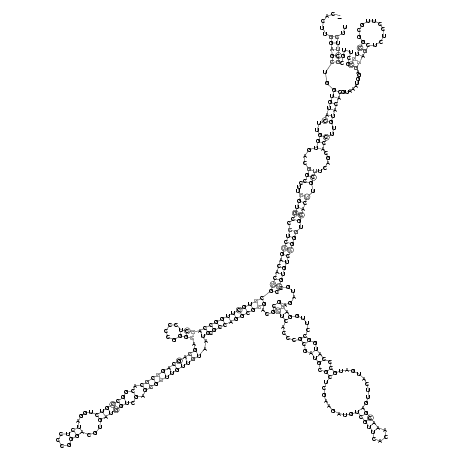
_CACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCACCCGCGAUGCGCUCGAAGAUGUCGUUCACAAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCCGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUAAAUGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
......(((((..((((((..(((...(((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.(.((((((....(((....)))...)))))).).)))))))))))..)).))))))))))...((((....((.(((.((.........(((((....))))).........)).))).))....))))......))))))))))).))))).))).....)))..)))))).........(((((..........)))))......))))).. (-93.67 = -96.35 + 2.68)
72961_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004