Index >
Results for CNB 81335
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
81335_ENSG00000179388_HUMAN_9329_9428/1-100 CTAATGTTCCATTGTGAGGAGCTTCCATTGTGACGTCGCGCCCCTTCGGCTGGGCTTTGT
81335_ENSDARG00000011592_ZEBRAFISH_9592_9685/1-100 -TAATGTTCCACTATGAGGAGCTTCCAT-GTGACGTCGCGCGGC--CGTCTAG-CTTTGT
81335_ENSRNOG00000017828_RAT_9150_9249/1-100 CTAATGTTCCATTGTGAGGAGCTTCCATTGTGACGTCGCGCCCCTTCGGCTGGGCTTTGT
81335_ENSMUSG00000033730_MOUSE_9335_9433/1-100 -TAATGTTCCATTGTGAGGAGCTTCCATTGTGACGTCGCGCCCCTTCGGCTGGGCTTTGT
********** * ************** ************ * ** ** * ******
81335_ENSG00000179388_HUMAN_9329_9428/1-100 CTGTCCATATATGGGCAGCTACGTCACGGAGCTTTCCCGG
81335_ENSDARG00000011592_ZEBRAFISH_9592_9685/1-100 GTGTCCATAAATGGTCATCTACGTCACGAAGTTGTTCCG-
81335_ENSRNOG00000017828_RAT_9150_9249/1-100 CTGTCCATATATGGGCAGCTACGTCACGGAGCTTTCCCGG
81335_ENSMUSG00000033730_MOUSE_9335_9433/1-100 CTGTCCATATATGGGCAGCTACGTCACGGAGCTTTCCCGG
******** **** ** ********** ** * * ***
//
81335.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 100
Mean pairwise identity: 89.82
Mean single sequence MFE: -35.53
Consensus MFE: -28.17
Energy contribution: -29.05
Covariance contribution: 0.88
Mean z-score: -1.88
Structure conservation index: 0.79
SVM decision value: 0.64
SVM RNA-class probability: 0.807337
Prediction: RNA
######################################################################
>81335_ENSG00000179388_HUMAN_9329_9428/1-100
CUAAUGUUCCAUUGUGAGGAGCUUCCAUUGUGACGUCGCGCCCCUUCGGCUGGGCUUUGUCUGUCCAUAUAUGGGCAGCUACGUCACGGAGCUUUCCCGG
.....(((((.......((.....))...(((((((.(((((((....)..))))......((((((....)))))))).))))))))))))........ ( -38.40)
>81335_ENSDARG00000011592_ZEBRAFISH_9592_9685/1-100
UAAUGUUCCACUAUGAGGAGCUUCCAUGUGACGUCGCGCGGCCGUCUAGCUUUGUGUGUCCAUAAAUGGUCAUCUACGUCACGAAGUUGUUCCG
................(((((..(..((((((((((((.(((......))).))))((.(((....))).))...))))))))..)..))))). ( -26.90)
>81335_ENSRNOG00000017828_RAT_9150_9249/1-100
CUAAUGUUCCAUUGUGAGGAGCUUCCAUUGUGACGUCGCGCCCCUUCGGCUGGGCUUUGUCUGUCCAUAUAUGGGCAGCUACGUCACGGAGCUUUCCCGG
.....(((((.......((.....))...(((((((.(((((((....)..))))......((((((....)))))))).))))))))))))........ ( -38.40)
>81335_ENSMUSG00000033730_MOUSE_9335_9433/1-100
UAAUGUUCCAUUGUGAGGAGCUUCCAUUGUGACGUCGCGCCCCUUCGGCUGGGCUUUGUCUGUCCAUAUAUGGGCAGCUACGUCACGGAGCUUUCCCGG
....(((((.......((.....))...(((((((.(((((((....)..))))......((((((....)))))))).))))))))))))........ ( -38.40)
>consensus
_UAAUGUUCCAUUGUGAGGAGCUUCCAUUGUGACGUCGCGCCCCUUCGGCUGGGCUUUGUCUGUCCAUAUAUGGGCAGCUACGUCACGGAGCUUUCCCGG
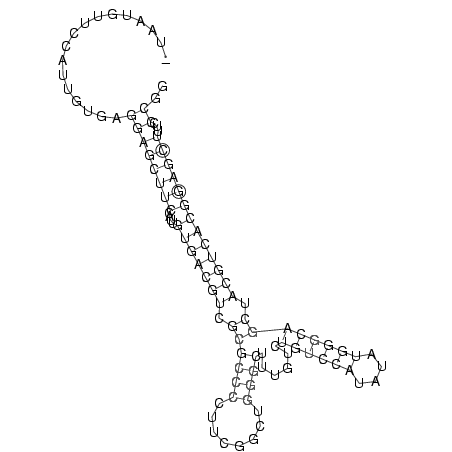
.................((((((((....(((((((.((((((........))))......((((((....)))))))).)))))))))))))...)).. (-28.17 = -29.05 + 0.88)
81335.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004